【論文】Phyuさん:Epidemiology and Genetic Analysis of SARS-CoV-2 in Myanmar during the Community Outbreaks in 2020
ミャンマーからの留学生Wint Wint Phyuさんの論文の紹介です
Phyu WW, Saito R, Wagatsuma K, Abe T, Tin HH, Pe EH, Win SMK, Win NC, Di Ja L, Tsuyoshi S, Makoto K, Kyaw Y, Chon I, Watanabe S, Hasegawa H, Watanabe H.
Epidemiology and Genetic Analysis of SARS-CoV-2 in Myanmar during the Community Outbreaks in 2020.
Viruses. 2022 Jan 27;14(2):259. doi: 10.3390/v14020259. PMID: 35215852; PMCID: PMC8875553.
ミャンマーで2020年に起こった新型コロナウイルスの地域流行とその遺伝子型についての報告です
Abstract
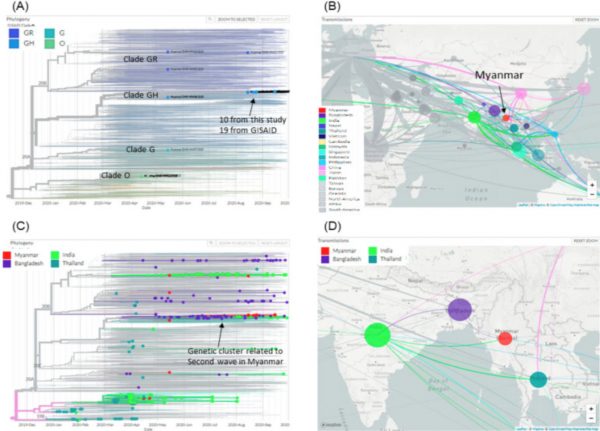
We aimed to analyze the situation of the first two epidemic waves in Myanmar using the publicly available daily situation of COVID-19 and whole-genome sequencing data of SARS-CoV-2. From March 23 to December 31, 2020, there were 33,917 confirmed cases and 741 deaths in Myanmar (case fatality rate of 2.18%). The first wave in Myanmar from March to July was linked to overseas travel, and then a second wave started from Rakhine State, a western border state, leading to the second wave spreading countrywide in Myanmar from August to December 2020. The estimated effective reproductive number (Rt) nationwide reached 6-8 at the beginning of each wave and gradually decreased as the epidemic spread to the community. The whole-genome analysis of 10 Myanmar SARS-CoV-2 strains together with 31 previously registered strains showed that the first wave was caused by GISAID clade O or PANGOLIN lineage B.6 and the second wave was changed to clade GH or lineage B.1.36.16 with a close genetic relationship with other South Asian strains. Constant monitoring of epidemiological situations combined with SARS-CoV-2 genome analysis is important for adjusting public health measures to mitigate the community transmissions of COVID-19.
公開されているCOVID-19の疫学情報とSARS-CoV-2の全ゲノムシークエンスデータを用いて、ミャンマーにおける最初の2回の流行波の状況を解析することを目的とした。2020年3月23日から12月31日までに、ミャンマーでは33,917人の患者が確認され、741人が死亡した(症例致死率2.18%)。3月から7月にかけてミャンマーで発生した第1波は海外渡航と関連し、その後、西部の国境州であるラカイン州から第2波が始まり、2020年8月から12月にかけてミャンマーの全国に第2波が広がりました。ミャンマーの実効再生産数(Rt)は各波の初期に6~8に達し、地域への流行の広がりとともに徐々に減少していきました。ミャンマーのSARS-CoV-2株10株と既登録株31株の全ゲノム解析から、第1波はGISAID clade OまたはPANGOLIN lineage B.6、第2波は他の南アジア株と遺伝的関係が深いclade GHまたは lineage B.1.36.16に変化したことが示された。SARS-CoV-2のゲノム解析と組み合わせて疫学的状況を常に監視することは、COVID-19の地域感染を軽減するための公衆衛生対策を調整するために重要である。