2025年3月14日 抗インフルエンザ剤感受性低下株調査の結果
2024年10月から2025年3月14日までの期間にわたり、日本各地でのインフルエンザ流行状況および薬剤耐性変異を持つインフルエンザウイルスの発生状況を調査しました。本調査では、バロキサビルに対する感受性が低下するPA遺伝子の変異、およびオセルタミビルを含むノイラミニダーゼ阻害剤に対する感受性が低下するNA遺伝子の変異を検出しました。
全国7道府県の外来医療機関が本研究に協力しました。発熱、咳、喉の痛み、だるさなど、インフルエンザが疑われる症状(インフルエンザ様疾患、ILI)がある患者のうち、迅速診断テスト(RDT)でインフルエンザA型またはB型陽性と判定された方から、上気道検体を採取し、これを初診時のサンプルとしました。また、バロキサビルを投与された患者には、3~7日後に再診をお願いし、その際に再診時の上気道検体を採取しました。これらの検体はすべて新潟大学に送付され、リアルタイムPCR(RT-PCR)法を用いてインフルエンザウイルスの検出、型/亜型の判定、PA変異およびNA変異の検出を、RT-PCR、サンガー法、次世代シーケンシング(NGS)を用いて行いました。
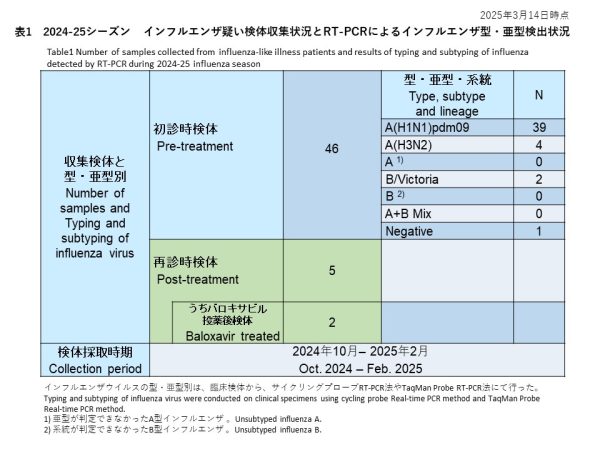
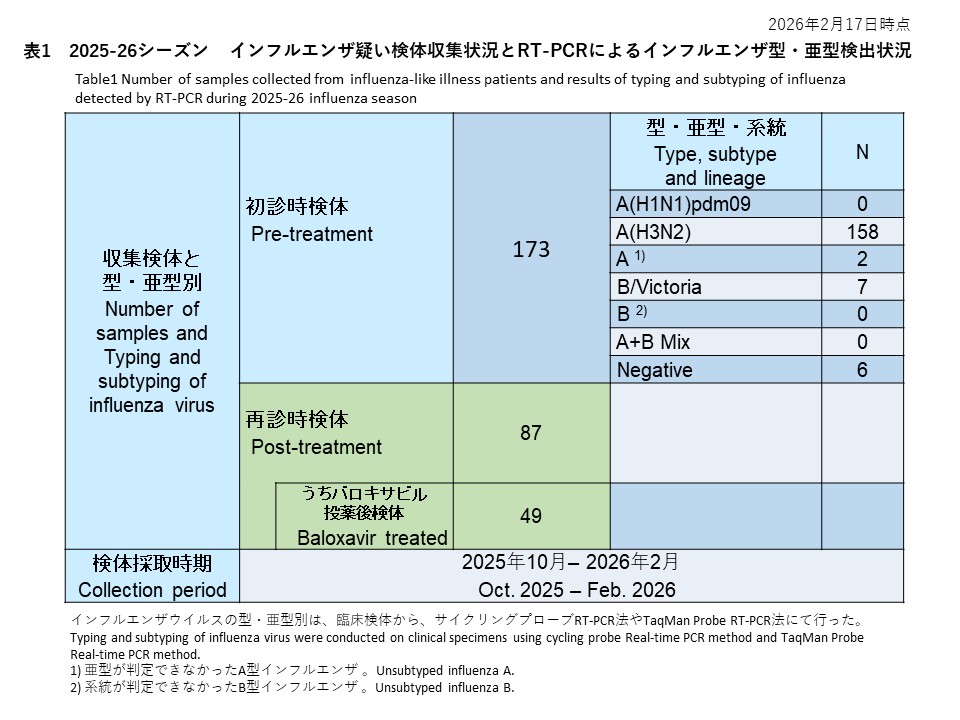
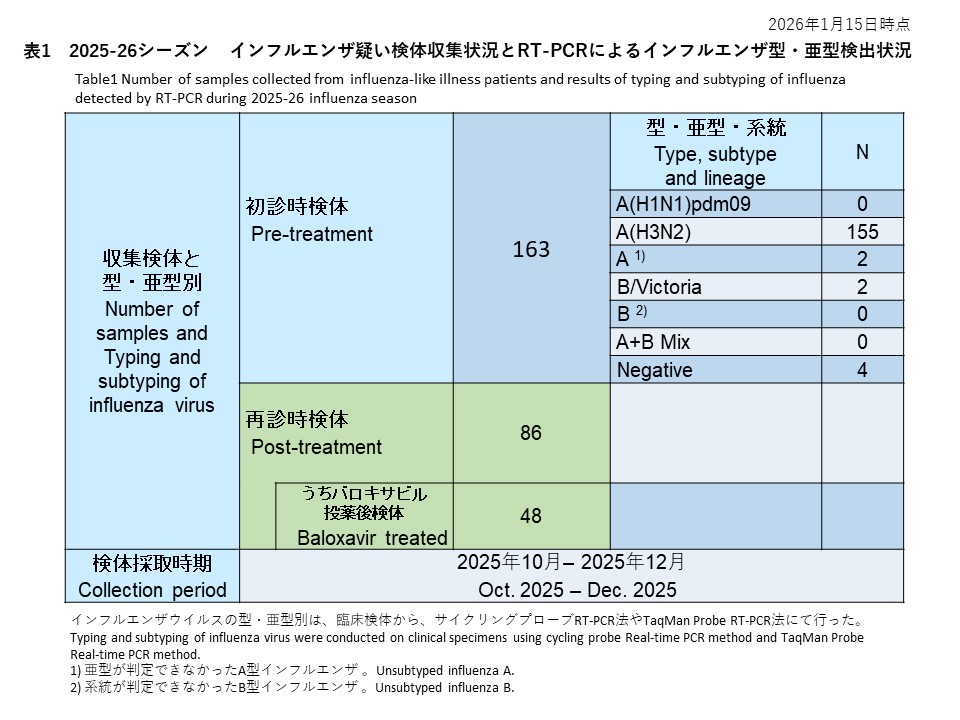
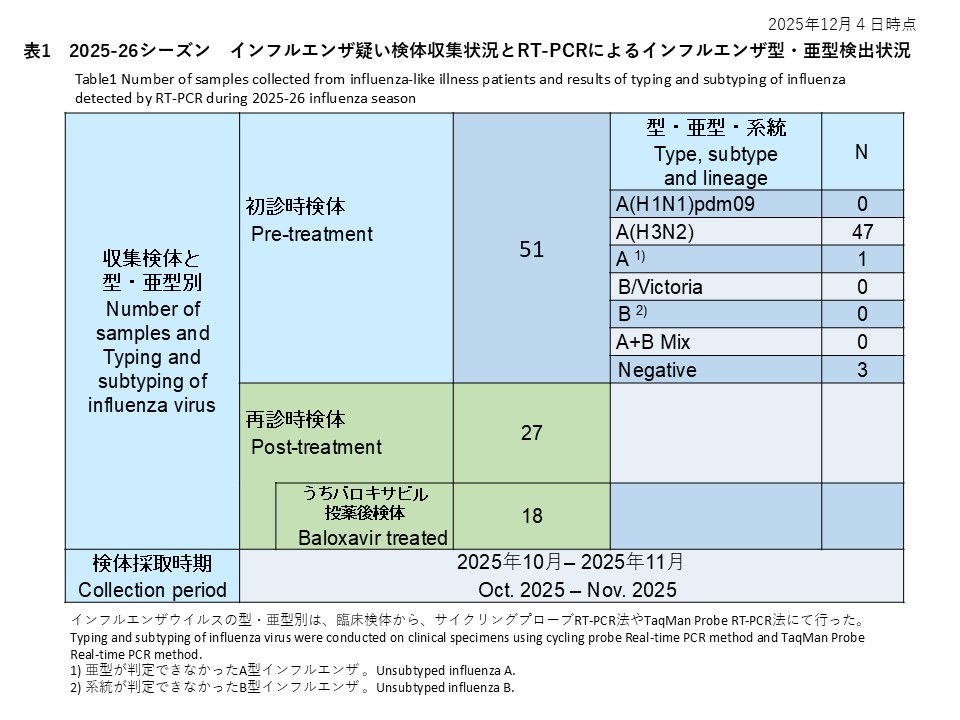
初診時に採取された46件の検体の内訳は、インフルエンザウイルスA(H1N1)pdm09が39件(84.8%)、インフルエンザウイルスA(H3N2)が4件(8.7%)、亜型判定不能のA型インフルエンザウイルスは0件(0.0%)、インフルエンザウイルスB/Victoriaが2件(4.3%)でした。また、A型およびB型の混合感染は0件(0.0%)、RT-PCR陰性の検体は1件(2.2%)でした(表1)。
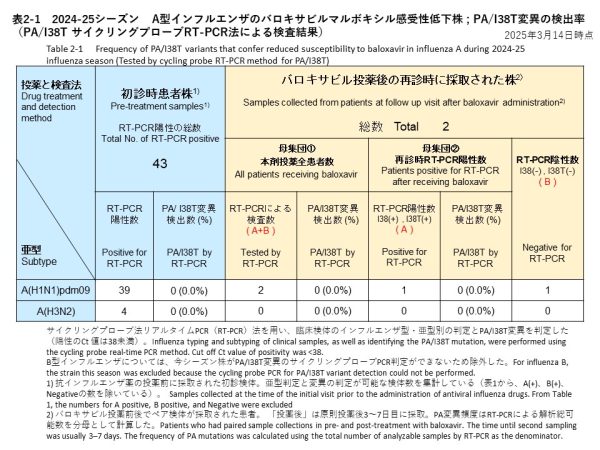
サイクリングプローブRT-PCR法を用いて、治療前に採取されたインフルエンザA型の検体から、PA/I38T変異の検出を試みた結果、39件のA(H1N1)pdm09と4件のA(H3N2)のいずれからもPA/I38T変異は確認されませんでした(検出率:0%)(表2-1)。
バロキサビル治療を受けた患者のうち2名(A(H1N1)pdm09が2名、A(H3N2)は0名)は、治療後3~7日目に再診した際、上気道検体を提供いただくことができました。再診時に採取された検体について、サイクリングプローブRT-PCR法を用いてPA/I38T変異を調べた結果、いずれの検体からもPA/I38T変異は検出されませんでした(検出率:0%)。検査結果の内訳は、1件がPA/I38T変異無し(感受性株)、1件がPCR陰性でした(表2-1)。
次世代シーケンサー(NGS)による全ゲノム解析を用いて、PA遺伝子およびNA遺伝子について、感受性低下を示す変異を調査しました。
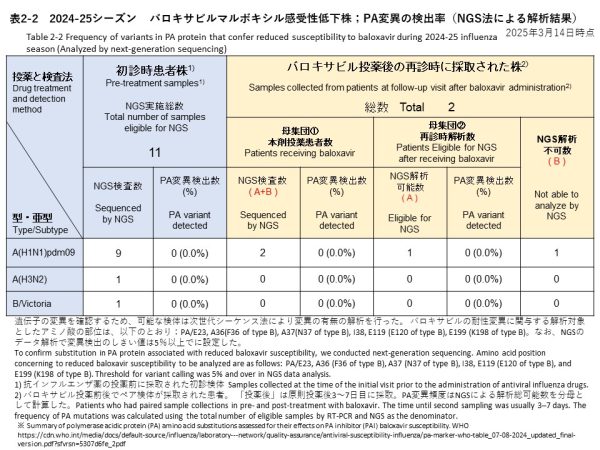
NGSで解析できた初診時検体は、11件でした。その内訳は、インフルエンザウイルスA(H1N1)pdm09が9件、インフルエンザウイルスA(H3N2)が1件、インフルエンザウイルスB/Victoriaが1件です(表2-2、表4)。
【PA遺伝子解析】
初診時に採取された検体において、バロキサビルに対する感受性低下を示すと報告された変異は確認されませんでした(表2-2)。
バロキサビル投与後の再診時に採取された検体のうち、A(H1N1)pdm09は1件、A(H3N2)は0件、B/Victoriaは0件、NGSにより解析することができました。
いずれの検体からもバロキサビルに対する感受性低下を示すと報告された変異は確認されませんでした。(検出率:0%)。
【NA遺伝子解析】
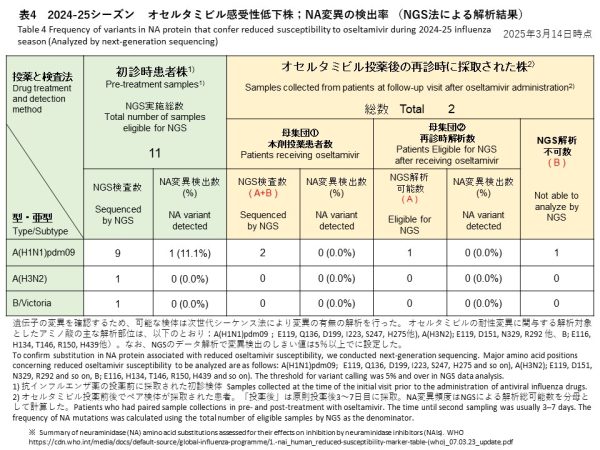
初診時に採取された検体のうち、オセルタミビルに対する感受性低下を示すと報告されているNA/N295S変異が、A(H1N1)pdm09で1件検出されました(検出率:11.1%)(表4)。バリアントコール(変異割合解析)の結果、この検体中に変異ウイルスが占める割合は17.9%でした。
その他の検体において、オセルタミビルに対する感受性低下を示すと報告された変異は確認されませんでした。
オセルタミビル投与後の再診時に採取された検体のうち、A(H1N1)pdm09は1件、A(H3N2)は0件、B Victoriaは0件、NGSにより解析することができました。
いずれの検体からもオセルタミビルに対する感受性低下を示すと報告されている変異は確認されませんでした(検出率:0%)。
なお、サンガー法を用いたPA遺伝子の変異検出は、現時点ではまだ実施されていません。
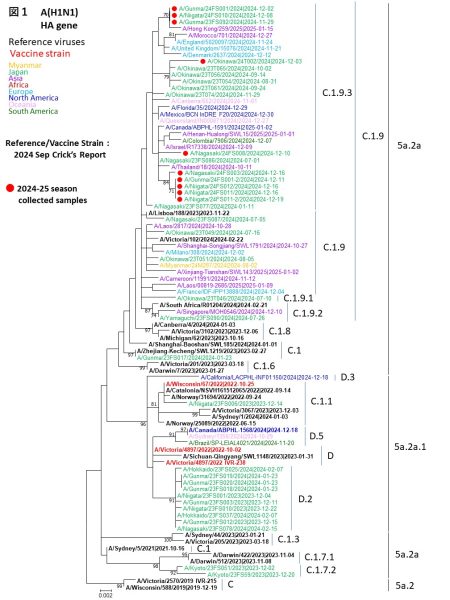
HA遺伝子による系統樹解析
2024年10月以降に日本で検出されたインフルエンザA(H1N1)pdm09のヘマグルチニン遺伝子(HA)の樹形図解析を実施いたしました(図1)。
今回解析した検体はすべて5a.2a(C.1.9.3)系統に分類されており(赤丸)、現在世界的に流行している株と一致していることが確認されました。
なお、2024-2025年シーズンのワクチン株(A/Victoria(ビクトリア) /4897/2022(IVR-238))に該当する5a.2a.1株は、現時点では日本ではまだ検出されておりません。
Overview of Influenza Circulating Strains and Antiviral Drug-Resistant Strains in Japan During the 2024-2025 Season (As of March 14, 2025)
Between October 2024 and March 14, 2025, we conducted a study across Japan to monitor influenza activity and the emergence of influenza virus strains with drug-resistant substitutions. This study aimed to detect substitutions in the PA gene, which reduce sensitivity to baloxavir, and substitutions in the NA gene, which reduce sensitivity to neuraminidase inhibitors, including oseltamivir.
Participating Medical Institutions and Sample Collection
Outpatient medical institutions in seven prefectures participated in this study. Upper respiratory tract samples were collected as initial samples from patients who visited these institutions with suspected influenza symptoms (influenza-like illness, ILI), such as fever, cough, sore throat, and fatigue. These patients were confirmed positive for influenza A or B using rapid diagnostic tests (RDTs). For patients treated with baloxavir, upper respiratory tract samples were also collected during follow-up visits 3–7 days after treatment. All clinical specimens were sent to Niigata University for influenza virus detection, subtype determination, and PA and NA substitution detection using real-time PCR(RT-PCR), Sanger sequencing, and next-generation sequencing (NGS).
Summary of Initial Sample Results
Among the 46 samples collected during the initial visit, 39 samples (84.8%) were identified as influenza A(H1N1)pdm09, while 4 samples (8.7%) were identified as influenza A(H3N2). No cases of unsubtypeable influenza A virus were detected (0.0%). Influenza B/Victoria was detected in two samples (4.3%). There were no cases of mixed A and B infections (0.0%). Additionally, one sample (2.2%) tested negative by RT-PCR (Table 1).
Detection of PA/I38T Substitution in Pre-Treatment Samples
Cycling probe RT-PCR was used to detect the PA/I38T substitution in pre-treatment influenza A samples. The substitution was not detected in any of the 39 A(H1N1)pdm09 or the 4 A(H3N2) samples analyzed, resulting in a detection rate of 0% (Table 2-1).
Follow-Up Samples After Baloxavir Treatment
Among the patients treated with baloxavir, two individuals, both of whom had A(H1N1)pdm09, returned for follow-up 3–7 days after treatment and provided upper respiratory tract samples. Cycling probe RT-PCR analysis revealed that neither of these samples carried the PA/I38T substitution, yielding a detection rate of 0%. Among the two samples, one was classified as a susceptible strain without the PA/I38T substitution, while the other tested negative by PCR (Table 2-1).
Whole-Genome Analysis Using Next-Generation Sequencing (NGS)
We conducted whole-genome analysis using NGS to investigate substitutions associated with reduced drug sensitivity in the PA and NA genes.
A total of 11 initial samples were successfully analyzed by NGS. Among these, nine samples were identified as A(H1N1)pdm09, one sample was A(H3N2), and one sample was B/Victoria (Tables 2-2 and 4).
PA Gene Analysis
In the initial samples, no substitutions previously reported to be associated with reduced sensitivity to baloxavir were detected (Table 2-2).
Among the follow-up samples collected after baloxavir treatment, NGS analysis was successfully conducted on one A(H1N1)pdm09 sample, while no A(H3N2) or B/Victoria samples were available for analysis. No substitutions associated with reduced sensitivity to baloxavir were detected in any of these samples, resulting in a detection rate of 0%.
NA Gene Analysis
Among the initial samples, the NA/N295S substitution, which has been reported to confer reduced sensitivity to oseltamivir, was detected in one A(H1N1)pdm09 sample, corresponding to a detection rate of 11.1% (Table 4). Variant calling analysis revealed that the proportion of substituted viruses within this sample was 17.9%. No other substitutions associated with reduced sensitivity to oseltamivir were detected in any of the other samples.
Among the follow-up samples collected after oseltamivir treatment, NGS analysis was successfully conducted on one A(H1N1)pdm09 sample, while no A(H3N2) or B/Victoria samples were available for analysis. No substitutions associated with reduced sensitivity to oseltamivir were detected in any of these samples, yielding a detection rate of 0%.
Pending Analyses
As of now, substitution detection in the PA gene using Sanger sequencing has not yet been conducted.
Phylogenetic Tree Analysis of HA gene
A phylogenetic tree analysis of the hemagglutinin (HA) gene of influenza A(H1N1)pdm09 detected in Japan since October 2024 was conducted (Figure 1). All the samples analyzed in this study belonged to the 5a.2a (C.1.9.3) lineage, which was confirmed to be consistent with strains currently circulating globally. The 5a.2a.1 strain, which is included in the 2024-2025 vaccine(vaccine strain : A/Victoria /4897/2022(IVR-238)), has not yet been detected in Japan as of now.