2025年12月4日 抗インフルエンザ剤感受性低下株調査の結果
2025-2026年シーズンの日本におけるインフルエンザ流行株と抗インフルエンザ薬感受性低下株調査の概要(2025年12月4日時点)
2025年10月から2025年12月4日までの期間にわたり、日本各地でのインフルエンザ流行状況および薬剤耐性変異を持つインフルエンザウイルスの発生状況を調査しました。本調査では、バロキサビルに対する感受性が低下するPA遺伝子の変異、およびオセルタミビルを含むノイラミニダーゼ阻害剤に対する感受性が低下するNA遺伝子の変異を検出しました。
全国8道府県の外来医療機関が本研究に協力しました。発熱、咳、喉の痛み、だるさなど、インフルエンザが疑われる症状(インフルエンザ様疾患、ILI)がある患者のうち、迅速診断テスト(RDT)でインフルエンザA型またはB型陽性と判定された方から、上気道検体を採取し、これを初診時のサンプルとしました。また、バロキサビルを投与された患者には、3~7日後に再診をお願いし、その際に再診時の上気道検体を採取しました。これらの検体はすべて新潟大学に送付され、リアルタイムPCR(RT-PCR)法を用いてインフルエンザウイルスの検出、型/亜型の判定、PA変異およびNA変異の検出を、RT-PCR、サンガー法、次世代シーケンシング(NGS)を用いて行いました。
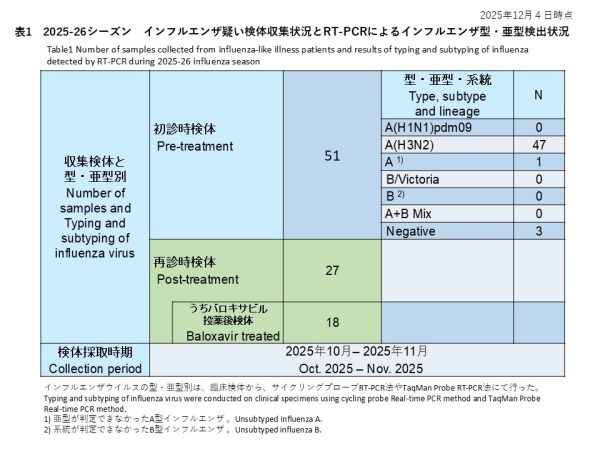
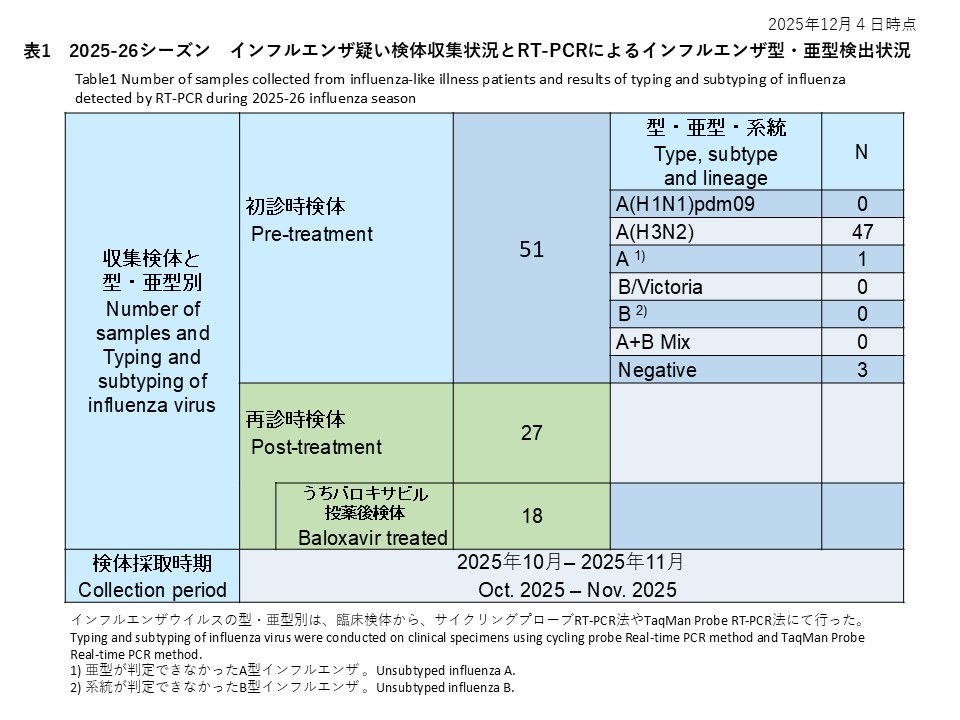
初診時に採取された51件の検体の内訳は、インフルエンザウイルスA(H1N1)pdm09が0件(0%)、インフルエンザウイルスA(H3N2)が47件(92.1%)、亜型判定不能のA型インフルエンザウイルスは1件(2.0%)、インフルエンザウイルスB/Victoriaが0件(0%)、系統が判定できなかったB型インフルエンザが0件(0%)でした。また、A型およびB型の混合感染は0件(0.0%)、RT-PCR陰性の検体は3件(5.9%)でした(表1)。
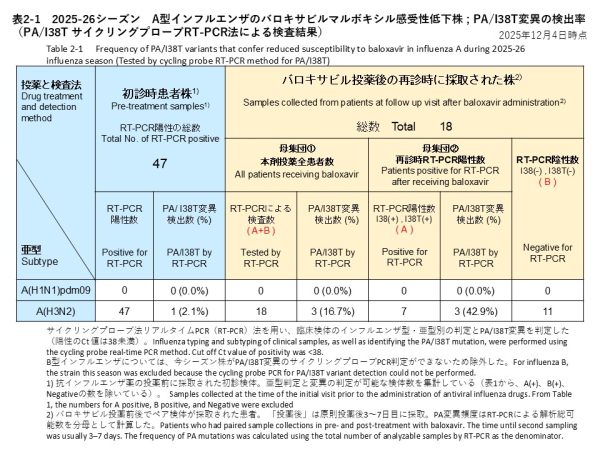
治療前に採取されたインフルエンザA型の検体について、サイクリングプローブRT-PCR法によりPA/I38T変異の検出を試みた結果、47件のA(H3N2)のうちPA/I38T変異陽性の検体が1検体確認されました(検出率:2.1%)(表2-1)。
バロキサビル治療を受けた患者のうち18名(全てA(H3N2)亜型)について、治療後3~7日目に再診時の上気道検体を提供いただきました。これらの検体に対して、サイクリングプローブRT-PCR法を用いてPA/I38T変異の有無を調べた結果、3検体からPA/I38T変異が検出されました(検出率:16.7%)。これら3検体は、変異のないウイルスも同時に検出されたため、バロキサビル感受性株と耐性変異株が混在していると考えられました。RT-PCR検査で陰性となった11検体を除く、陽性となった7検体を分母とした場合には、PA/I38T変異がある検体の割合は42.9%(3/7)となりました(表2-1)。これらの3検体が採取された患者の解熱時間や症状回復時間の詳細は確認中です。
なお、サンガー法を用いたPA遺伝子の変異検出や、NGS法によるPA遺伝子およびNA遺伝子の変異解析については、現時点ではまだ実施していません。
Overview of Influenza Circulating Strains and Antiviral Drug-Resistant Strains in Japan During the 2025-2026 Season (As of December 4 , 2025)
Between October 2025 and December 4, 2025, we conducted a study in Japan to monitor circulating influenza strains and the emergence of antiviral resistance. The study focused on detecting substitutions in the PA gene associated with reduced susceptibility to baloxavir and substitutions in the NA gene associated with reduced susceptibility to neuraminidase inhibitors, including oseltamivir.
Participating Medical Institutions and Sample Collection
Outpatient medical institutions in eight prefectures participated in this study. Upper respiratory tract samples were collected as initial samples from patients who visited these institutions with suspected influenza symptoms (influenza-like illness, ILI), such as fever, cough, sore throat, and fatigue. These patients were confirmed positive for influenza A or B using rapid diagnostic tests (RDTs). For patients treated with baloxavir, upper respiratory tract samples were also collected during follow-up visits 3–7 days after treatment. All clinical specimens were sent to Niigata University for influenza virus detection, subtype determination, and PA and NA substitution detection using real-time PCR(RT-PCR), Sanger sequencing, and next-generation sequencing (NGS).
Initial Sample Results
Of the 51 samples collected at the initial visit, 0 (0%) were identified as influenza A(H1N1)pdm09, 47 (92.1%) as A(H3N2), 0 (0%) as B/Victoria, and 0 (0%) as influenza B of undetermined lineage. No A/B co-infections were detected. Three samples (5.9%) were RT-PCR negative (Table 1).
Detection of PA/I38T Substitution in Pre-Treatment Samples
Cycling probe RT-PCR was performed to detect the PA/I38T substitution in influenza A samples collected prior to treatment. PA/I38T substitution was detected in one of 47 A(H3N2) samples (detection rate: 2.1%) (Table 2-1).
Follow-Up Samples After Baloxavir Treatment
Among 18 patients treated with baloxavir (all infected with influenza A(H3N2)), upper respiratory tract samples were collected 3–7 days post-treatment. Using a cycling probe RT-PCR assay, we examined these specimens for the presence of the PA/I38T substitution and detected PA/I38T in 3 specimens (detection rate: 16.7%). In all three specimens, viruses without the PA/I38T substitution were also detected, suggesting a mixed population of baloxavir-susceptible viruses and viruses carrying the resistance-associated substitution. When the 11 RT-PCR–negative specimens were excluded and the seven RT-PCR–positive specimens that could be subtyped were used as the denominator, the proportion of specimens with PA/I38T was 42.9% (3/7) (Table 2-1). Details of the fever duration and symptom duration of the patients from whom these three samples were collected are currently being confirmed.
Pending Analyses
PA gene substitution detection using Sanger sequencing and PA/NA substitution analysis using NGS has not yet been conducted.