2026年1月15日 抗インフルエンザ剤感受性低下株調査の結果
2025-2026年シーズンの日本におけるインフルエンザ流行株と抗インフルエンザ薬感受性低下株調査の概要(2026年1月15日時点)
2025年10月から2026年1月15日までの期間にわたり、日本各地でのインフルエンザ流行状況および薬剤耐性変異を持つインフルエンザウイルスの発生状況を調査しました。本調査では、バロキサビルに対する感受性が低下するPA遺伝子の変異、およびオセルタミビルを含むノイラミニダーゼ阻害剤に対する感受性が低下するNA遺伝子の変異を検出しました。
全国8道府県の外来医療機関が本研究に協力しました。発熱、咳、喉の痛み、だるさなど、インフルエンザが疑われる症状(インフルエンザ様疾患、ILI)がある患者のうち、迅速診断テスト(RDT)でインフルエンザA型またはB型陽性と判定された方から、上気道検体を採取し、これを初診時のサンプルとしました。また、バロキサビルを投与された患者には、3~7日後に再診をお願いし、その際に再診時の上気道検体を採取しました。これらの検体はすべて新潟大学に送付され、リアルタイムPCR(RT-PCR)法を用いてインフルエンザウイルスの検出、型/亜型の判定、PA/I38T変異の検出(A(H1N1)pdm09とA(H3N2)に対し)、NA/H275Y変異の検出(A(H1N1)pdm09に対し)を行いました。次世代シーケンシング(NGS)法とサンガー法を用いて、PA変異及びNA変異を確認しました。
【初診時検体】
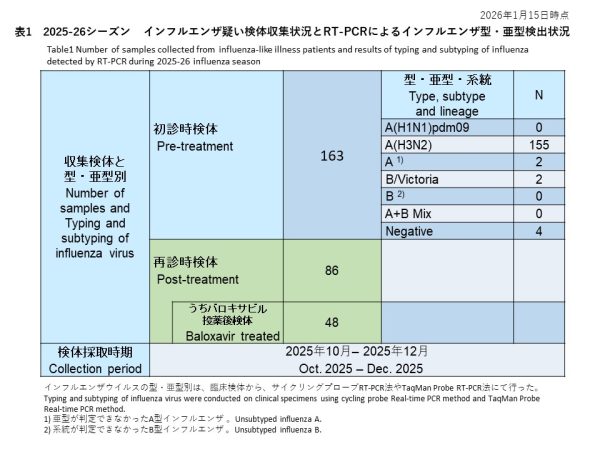
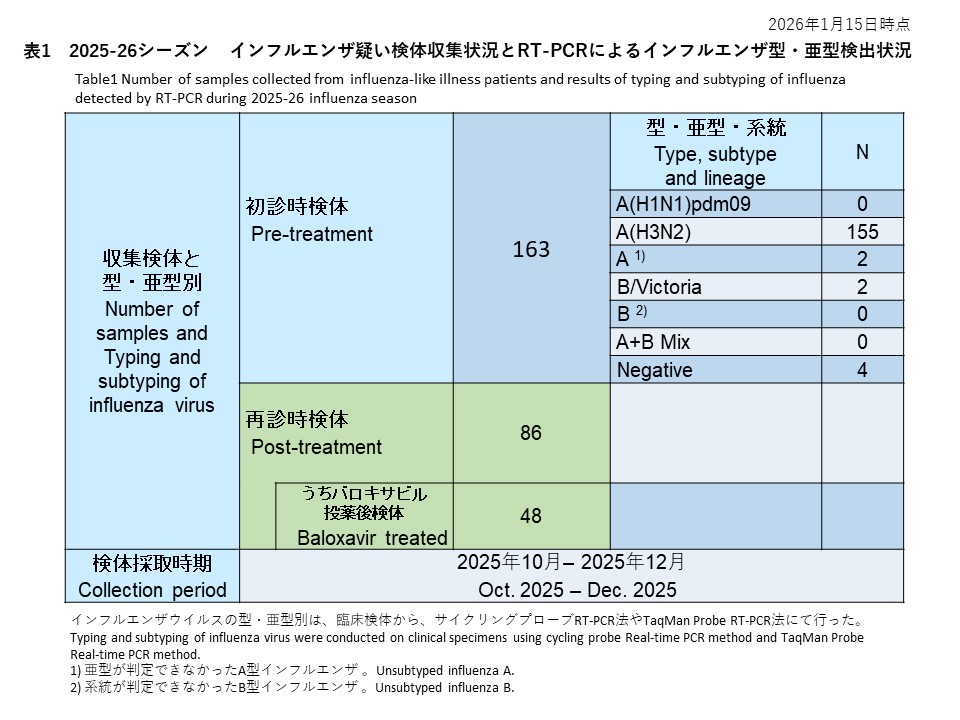
初診時に採取された163件の検体の内訳は、インフルエンザウイルスA(H1N1)pdm09が0件(0%)、インフルエンザウイルスA(H3N2)が155件(95.1%)、亜型判定不能のA型インフルエンザウイルスは2件(1.2%)、インフルエンザウイルスB/Victoriaが2件(1.2%)、系統が判定できなかったB型インフルエンザが0件(0%)でした。また、A型およびB型の混合感染は0件(0.0%)、RT-PCR陰性の検体は4件(2.5%)でした(表1)。
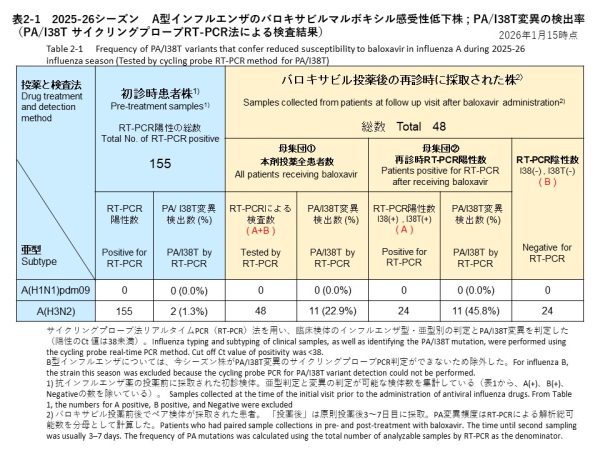
治療前に採取されたインフルエンザA型の検体について、サイクリングプローブRT-PCR法によりPA/I38T変異の検出を試みた結果、155件のA(H3N2)のうちPA/I38T変異陽性の検体が2検体確認されました(検出率:1.3%)(表2-1)。
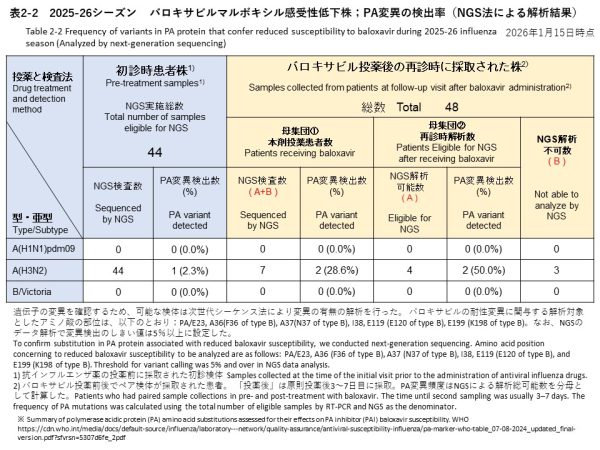
次世代シークエンス解析は、初診時検体155件のうちA(H3N2) 44件を対象に実施しました。その結果、バロキサビル耐性に関連するPA/I38T変異を1件で検出しました(検出率:2.3%)(表2-2)。当該検体のバリアントコール解析では、PA/I38T変異の割合が99%以上であり、野生型はほとんど検出されず、PA/I38T変異ウイルスがほぼ全てを占める状態でした。なお、この症例は、治療はバロキサビルでしたが、臨床経過の詳細は確認できていません。また、この症例の再診検体が採取されましたが、ウイルス量が少なく解析できませんでした。サイクリングプローブ法による初診時検体の検査で、PA/I38T変異陽性となったもう1件は未だNGS解析を行っていません。
また、初診時のA(H3N2)検体1件で、NGSバリアントコール(変異検出閾値5%)によりPA/E23Vが5.7%検出されました。E23Vは耐性関連変異リスト(例:E23G/K/R)に含まれず文献報告も確認できないため、参考所見として記載します(表2‐2には感受性株として計上)。
サンガー法によるPA遺伝子変異の解析は、現時点では未実施です。
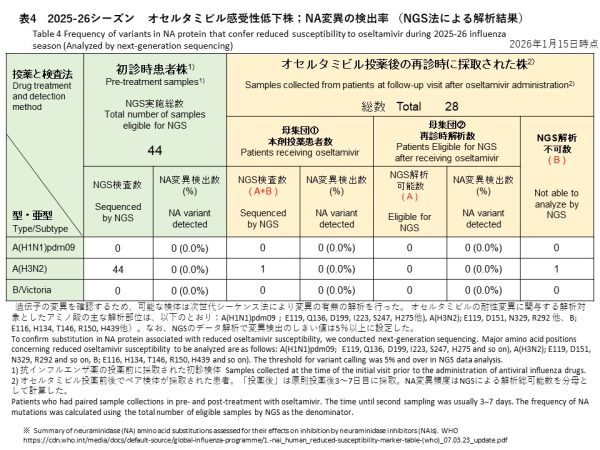
44件のA(H3N2)から、オセルタミビルに対する感受性低下に関連するNA遺伝子変異は、次世代シークエンス法で検出されませんでした(表4)。
【再診時検体】
再診時検体は86名から採取され、このうちバロキサビル治療を受けた患者48名(全てA(H3N2)亜型)について、治療後3~7日目に再診時の上気道検体を提供いただきました(表1)。
これらの検体に対して、サイクリングプローブRT-PCR法を用いてPA/I38T変異の有無を調べた結果、48検体中11検体でPA/I38T変異が検出されました(検出率:22.9%)。このうち1検体は、PA/I38T変異のあるウイルスのみが検出され、他10検体は変異のないウイルスも同時に検出されたため、バロキサビル感受性株と耐性変異株が混在していると考えられました。また、再診時にRT-PCRでインフルエンザA(H3N2)が陽性であった24検体に限ると、PA/I38T変異は11検体で検出され、検出割合は45.8%(11/24)でした(表2-1)。これらの11例のうち、4例の症状の経過が確認できており、有熱時間はそれぞれ、0時間、7時間、105時間、133時間でした。他の患者の有熱時間や有症状期間の詳細は確認中です。
また、1症例については再診の1日後にもう一回検体が採取されており(再々診)、再診検体ではバロキサビル感受性株と耐性変異株が混在していましたが、再々診検体では、PA/I38Tの耐性変異株のみが検出されました。
86件の再診時検体のうち、NGS検査を実施したのは8件で、そのうち7件がバロキサビル投与後、1件がオセルタミビル投与後の検体でした(表2-2、表4)。このバロキサビル投与後の7検体(全てA(H3N2))のNGS解析では、4件でデータ解析が可能で、PA遺伝子の変異は4件中2件で確認されました。検出された変異はいずれもPA/I38Tで、バリアントコール頻度はそれぞれ20.8%、54.7%でした。PA変異の検出率は解析対象7件を分母とすると28.6%(2/7)、解析が成功した4件を分母とすると50.0%(2/4)でした。
オセルタミビル投与後の再診時に採取されたA(H3N2)検体1件についてNGS解析を実施しましたが、解析に必要なデータが得られませんでした。
Overview of Influenza Circulating Strains and Antiviral Drug-Resistant Strains in Japan During the 2025-2026 Season (As of January 15, 2026)
Between October 2025 and January 15, 2026, we conducted a study in Japan to monitor circulating influenza strains and the emergence of antiviral resistance. The study focused on detecting substitutions in the PA gene associated with reduced susceptibility to baloxavir and substitutions in the NA gene associated with reduced susceptibility to neuraminidase inhibitors, including oseltamivir.
Participating Medical Institutions and Sample Collection
Outpatient medical institutions in eight prefectures participated in this study. Upper respiratory tract samples were collected as initial samples from patients who visited these institutions with suspected influenza symptoms (influenza-like illness, ILI), such as fever, cough, sore throat, and fatigue. These patients were confirmed positive for influenza A or B using rapid diagnostic tests (RDTs). For patients treated with baloxavir, upper respiratory tract samples were also collected during follow-up visits 3–7 days after treatment. All clinical specimens were sent to Niigata University, where real-time RT-PCR was performed for influenza virus detection, type/subtype determination, detection of the PA/I38T substitution (for A(H1N1)pdm09 and A(H3N2)), and detection of the NA/H275Y substitution (for A(H1N1)pdm09). Next-generation sequencing (NGS) and Sanger sequencing were used to confirm PA and NA substitutions.
Baseline (pre-treatment) specimens
Among 163 baseline specimens, influenza A(H1N1)pdm09 was detected in 0 specimens (0%), influenza A(H3N2) in 155 specimens (95.1%), unsubtyped influenza A in 2 specimens (1.2%), influenza B/Victoria in 2 specimens (1.2%), and influenza B with undetermined lineage in 0 specimens (0%). No mixed A and B infections were identified (0.0%), and 4 specimens (2.5%) were RT-PCR negative (Table 1).
For baseline pre-treatment influenza A specimens, we screened for the PA/I38T substitution using the cycling-probe RT-PCR method. PA/I38T-positive specimens were identified in 2 of 155 A(H3N2) specimens (detection rate: 1.3%) (Table 2-1).
NGS analysis was performed for 44 of the 155 baseline A(H3N2) specimens. As a result, the PA/I38T substitution associated with reduced susceptibility to baloxavir was detected in 1 specimen (detection rate: 2.3%) (Table 2-2). Variant-calling analysis showed that the PA/I38T variant frequency was ≥99%, indicating that wild-type virus was barely detectable and that the PA/I38T variant population accounted for almost the entire viral population in the specimen. For this case, baloxavir was used for treatment; however, detailed clinical course information could not be confirmed. A follow-up specimen was collected from this case, but the viral load was low and sequencing analysis was not successful. For the other baseline specimen that was PA/I38T-positive by the cycling-probe method, NGS analysis has not yet been performed.
In addition, in one baseline A(H3N2) specimen, PA/E23V was detected at 5.7% by NGS variant calling (variant detection threshold: 5%). Because E23V is not included in the list of baloxavir resistance-associated substitutions by WHO (e.g., E23G/K/R) and no published reports have been identified, this finding is described as a reference (this sample is counted as a susceptible virus in Table 2-2).
Sanger sequencing for PA gene substitutions has not been performed to date.
Among the 44 baseline A(H3N2) specimens analyzed by NGS, no NA gene substitutions associated with reduced susceptibility to oseltamivir were detected (Table 4).
Follow-up (post-treatment) specimens
Follow-up (post-treatment) specimens were collected from 86 patients. Among them, 48 patients, all A(H3N2), had a follow up sample collection 3–7 days after the start of baloxavir treatment (Table 1).
Using the cycling-probe RT-PCR method, PA/I38T was detected in 11 of the 48 follow-up specimens (detection rate: 22.9%). In one specimen, only the PA/I38T variant virus was detected, whereas in the remaining 10 specimens, wild-type virus was also detected, suggesting mixed populations of baloxavir-susceptible virus and resistance-associated variants. When analysis was restricted to the 24 follow-up specimens that were RT-PCR positive for influenza A(H3N2), PA/I38T was detected in 11 specimens, corresponding to a detection proportion of 45.8% (11/24) (Table 2-1). Among these 11 cases, symptom courses were available for 4 cases, with fever duration of 0 hours, 7 hours, 105 hours, and 133 hours, respectively. Detailed information on fever duration and symptom duration for the remaining patients is under confirmation.
In one case, an additional specimen (a second follow-up visit) was collected one day after the follow-up visit. While the first follow-up specimen contained a mixed population of baloxavir-susceptible virus and PA/I38T variant virus, only the PA/I38T variant virus was detected in the second follow-up specimen.
Among the 86 follow-up specimens, NGS was performed for 8 specimens: 7 specimens collected after baloxavir treatment and 1 specimen collected after oseltamivir treatment (Tables 2-2 and 4). For the 7 post-baloxavir A(H3N2) specimens, sequencing analysis was successful in 4 specimens, and PA gene substitutions were identified in 2 of these 4 specimens. The detected substitutions were PA/I38T in both specimens, with variant frequencies of 20.8% and 54.7%, respectively. The detection rate of PA substitutions was 28.6% (2/7) when calculated using all 7 specimens as the denominator, and 50.0% (2/4) when calculated using the 4 specimens with successful analysis as the denominator.
For the single follow-up A(H3N2) specimen collected after oseltamivir treatment, NGS was performed; however, no sufficient data for analysis were obtained.