2024年3月21日 抗インフルエンザ剤感受性低下株調査の結果
結果の概要
2024年3月21日時点
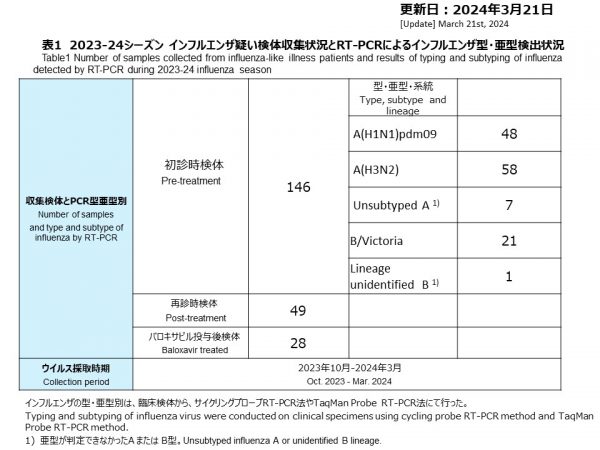
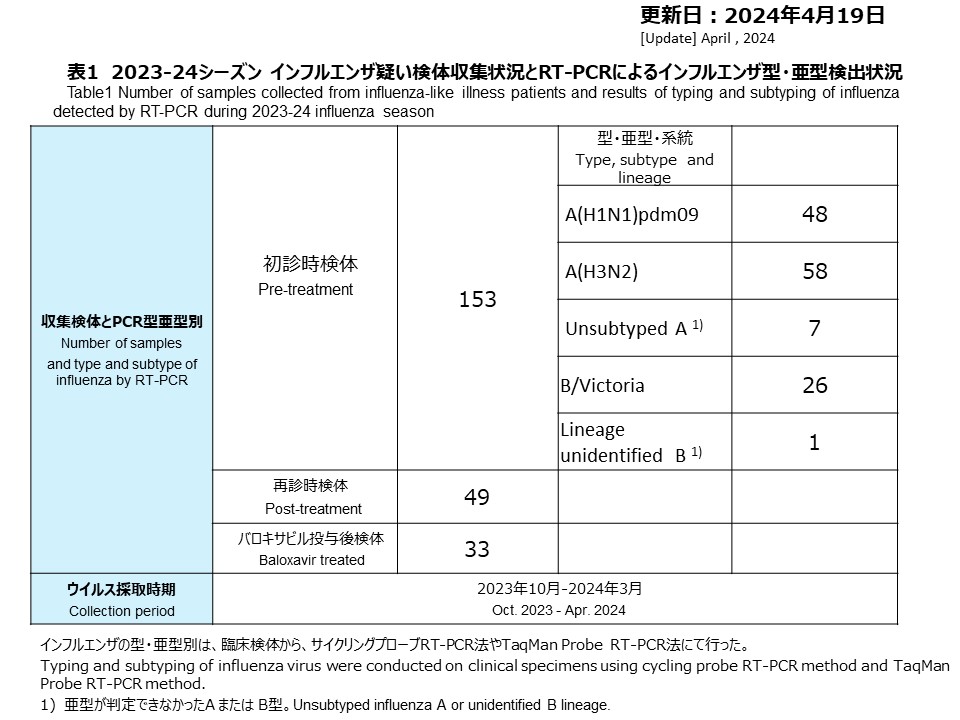
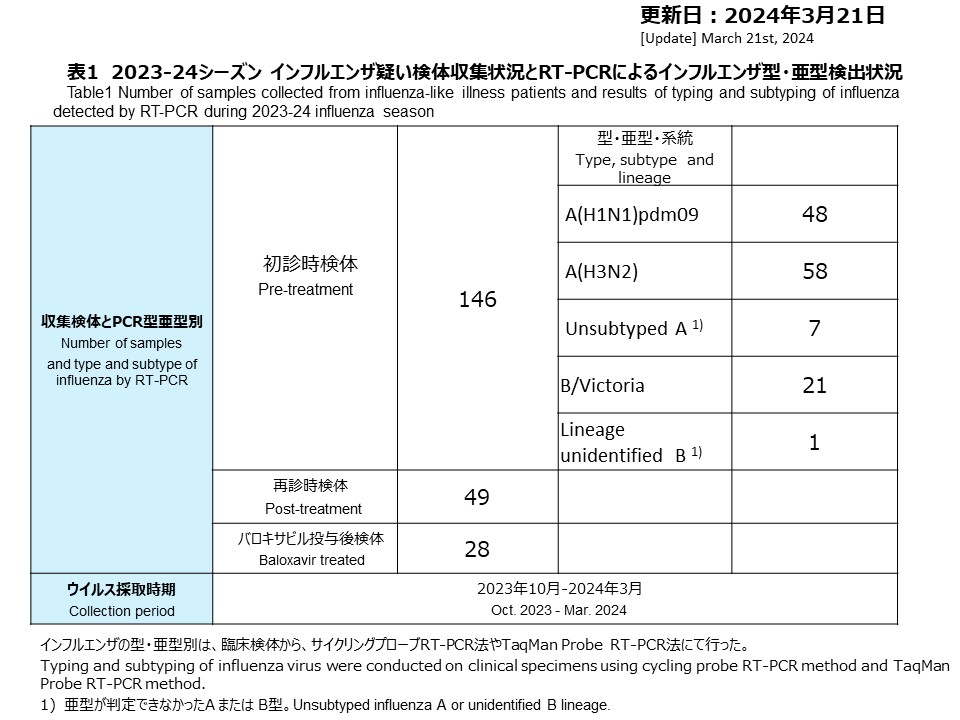
2023年10月から2024年2月にかけて、合計146件の初診時検体が採取されました。再診時検体は49件採取され、うち28件はバロキサビル投与後に採取されました(表1)。
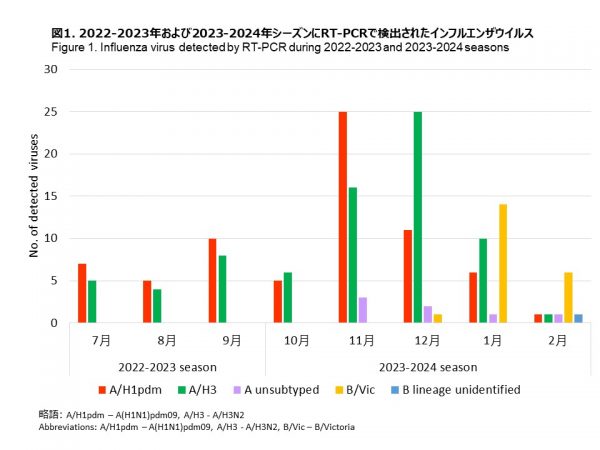
2023年10月以降、インフルエンザA(H1N1)pdm09を48件、A(H3N2)を58件、インフルエンザA型(亜型が特定不能)を7件、B/ビクトリアを21件、インフルエンザB型(系統が特定不能)を1件検出しました(表1,図1)。今シーズンはA(H1N1)pdm09とA(H3N2)の同時流行であり、インフルエンザ検出数は11月が最も多く、12月以降は徐々に減っています。
インフルエンザウイルスのPA蛋白38位がイソロイシンからスレオニンに変異したウイルス(PA/I38T変異株)は、バロキサビルに対して感受性低下を示すと言われています。このPA/I38T変異株を特異的に検出するリアルタイムPCR法(サイクリンブプローブ法)を採取された検体全てに実施しました。
今回特筆すべき結果として、リアルタイムPCRおよび次世代シーケンシングにより、バロキサビル治療後にPA/I38T変異を有するインフルエンザA(H3N2)ウイルスを1株検出しました。
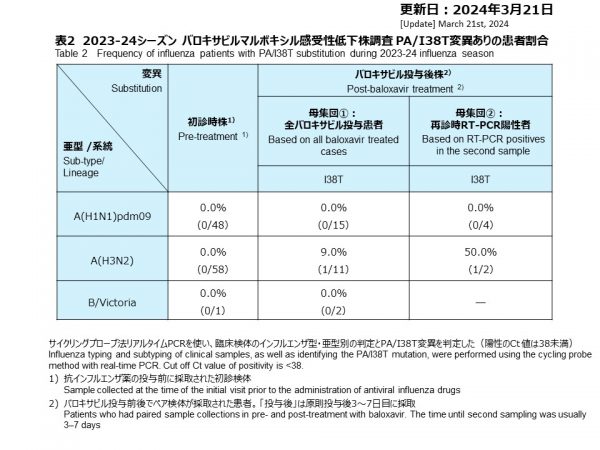
リアルタイムPCR法により、PA/I38T変異を検出したところ、抗インフルエンザ薬投与前のA(H1N1)pdm09の48株、A(H3N2)の58株、B型の1株のいずれにおいてもPA/I38T変異は検出されませんでした(表2)。
バロキサビル投与後に採取された検体(再診時検体)は28件でした(表2)。15件がA(H1N1)pdm09で、うち4件がPCR陽性でしたが、PA/I38T変異は検出されませんでした。A(H3N2)は11件で、2件がPCR陽性で、うち1件にPA/I38T変異が検出され、この出現頻度は9.0%でした( 1/11)。B/Victoriaは2件でしたが、再診のPCR結果では全て陰性でした。
[PA遺伝子解析]
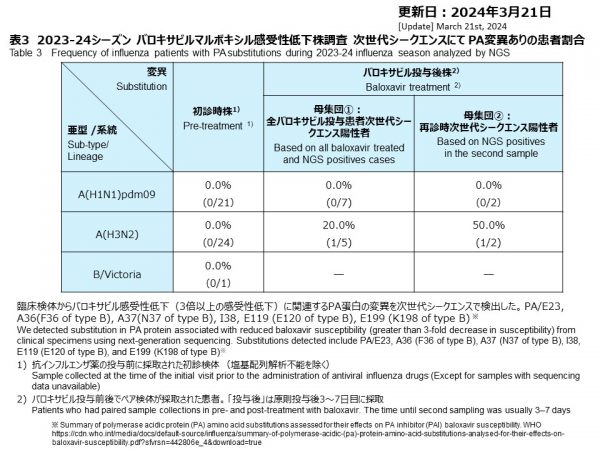
次世代シークエンシング(NGS)を実施した初診時検体46件は、A(H1N1)pdm09が21件、A(H3N2)が24件、B型が1件で、バロキサビルに対する感受性低下を示すと報告されたPA変異はすべての検体にありませんでした。
バロキサビル投与後に採取された検体(再診時検体)は、A(H1N1)pdm09が2件、A(H3N2)が2件でした。A(H1N1)pdm09にはバロキサビルに対する感受性低下を示すと報告された変異は検出されませんでしたが(0.0%, 0/2)、A(H3N2)の1件ではPA/I38T変異が検出されました(20.0%, 1/5)(表3)。
[NA遺伝子解析]
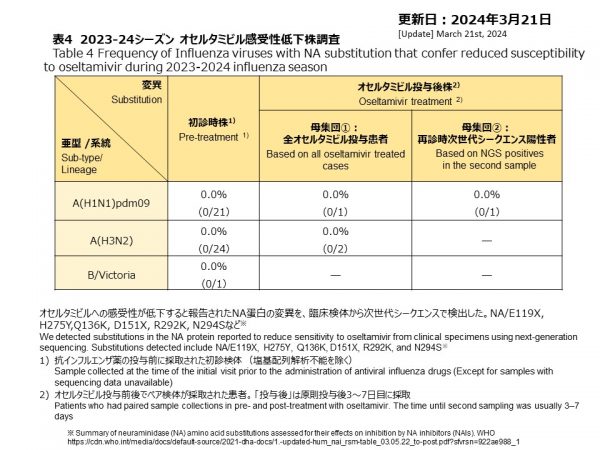
変異について、NGSを実施した初診時検体46検体は、オセルタミビルを含むNA阻害剤に対する感受性低下を示すと報告されたNA変異はすべての検体にありませんでした。オセルタミビル投与後に採取された検体A(H1N1)pdm09の1件、A(H3N2)の2件、B型の1件には、NA阻害剤に対する感受性を低下させる変異はNA遺伝子に検出されませんでした(表4)。
Summary of Results.
As of March 21, 2024
From October 2023 to February 2024, a total of 146 initial visit specimens were collected. There were 49 specimens collected at follow-up visits (second visits), of which 28 were collected after administration of baloxavir (Table 1).
Since October 2023, 48 cases of influenza A(H1N1)pdm09, 58 cases of A(H3N2), 7 cases of influenza A (subtype unidentified), 21 case of B/Victoria and 1 case of B lineage unidentified were detected (Table 1 and Figure 1). This season saw a simultaneous circulation of A(H1N1)pdm09, A(H3N2) and B/Victoria with the highest number of influenza detections in November, gradually decreasing from December onwards.
A virus with a substitution at position 38 of the PA protein, from isoleucine to threonine (PA/I38T mutation), is reported to confer reduced sensitivity to baloxavir. We implemented a real-time PCR method (cycling probe method) specifically designed to detect this PA/I38T mutation on all samples.
Of note, we found one influenza A(H3N2) virus with PA/I38T substitution after the treatment of baloxavir by real-time PCR and next generation sequencing.
None of the 48 of A(H1N1)pdm09, 58 of A(H3N2), and 21 of influenza B/Victoria collected before administration of antiviral influenza drugs showed the presence of the PA/I38T mutation by real-time PCR (Table 2).
There were 28 specimens collected after administration of baloxavir (follow-up visit specimens) (Table 2). Of these, 15 were A(H1N1)pdm09, with 4 testing positive via PCR, and the PA/I38T substitution was not detected. Among the 11 cases of A(H3N2), one tested positive for PA/I38T. This mutation appeared at a frequency of 9.0% (1/11). There were two B/Victoria cases, which were PCR negative at the 2nd visit.
[PA Gene Analysis]
Forty-six initial visit specimens were subjected to next-generation sequencing (NGS), with 21 cases of A(H1N1)pdm09, 24 case of A(H3N2), and 1 case of type B. None of first visit specimen had any PA substitution, with reduced susceptibility to baloxavir. Two influenza A(H1N1)pdm09 and two A(H3N2) post-baloxavir-treated viruses underwent NGS. No PA-variants was founded among A(H1N1)pdm09 strains (0.0%, 0/2). One influenza A(H3N2)-infected, post-baloxavir-treated case, with PA/I38T substitution was confirmed in the PA protein by NGS (20%, 1/5) (Table 3).
[NA Gene Analysis]
Same 46 strains analyzed for NA substitutions, affecting sensitivity to neuraminidase inhibitors (NAIs). NGS results showed no substitution in the NA gene affecting sensitivity to NAIs in the 46 prior to treatment cases of A(H1N1)pdm09, A(H3N2) and B/Victoria (Table 4). One influenza A(H1N1)pdm09 and 2 A(H3N2) post-oseltamivir-treated sample were sequenced and no any variants, inhibiting NAIs was confirmed (Table 4).