2023-2024年シーズンの日本におけるインフルエンザサーベイランスの概要(2024年12月25日作成)
2023-2024年シーズンの日本におけるインフルエンザ流行株と抗インフルエンザ薬感受性低下株調査の概要(2024年12月25日作成)
2023年10月から2024年9月までの期間中、日本各地でインフルエンザの流行動向とバロキサビルおよびオセルタミビルに対する薬剤耐性変異を持つインフルエンザウイルスの発生状況について調べました。
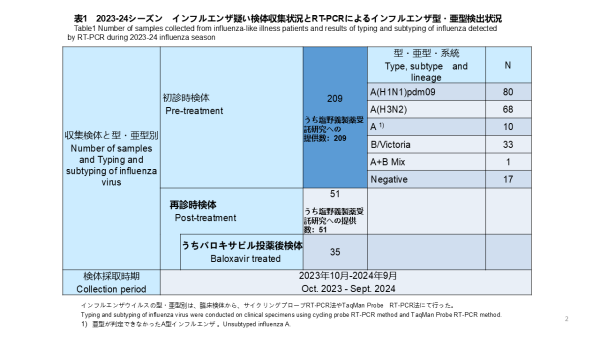
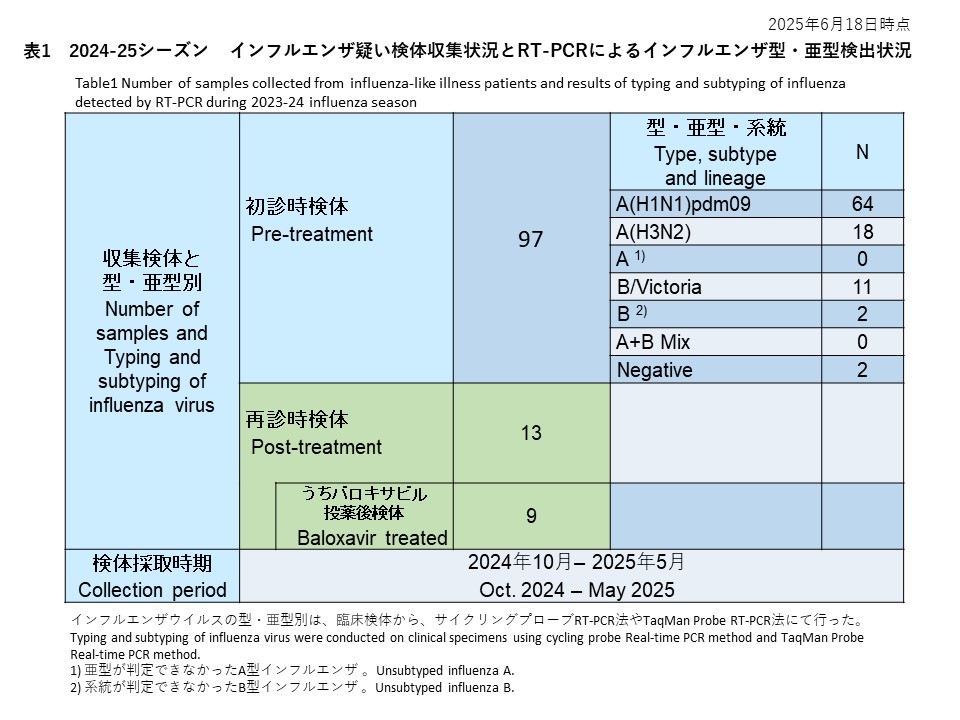
全国8つの外来医療機関が研究に参加しました。医療機関を受診し、迅速診断テストキット(RDT)で陽性と判定されたインフルエンザ様疾患(ILI)患者から、上気道スワブを検体として収集しました。これらの臨床検体は全て新潟大学に送られ、RT-PCR法でインフルエンザウイルスの検出と型/亜型の決定を行いました。採取された209件の検体のうち、80件(38.3%)がインフルエンザウイルスA(H1N1)pdm09、68件(32.5%)がインフルエンザウイルスA(H3N2)、10件(4.8%)が亜型判定不能のインフルエンザウイルスA型、33件(15.8%)がインフルエンザウイルスB/Victoria、A型およびB型が混合感染していたものが1件(0.5%)、17件(8.1%)がRT-PCRで陰性でした(表1)。
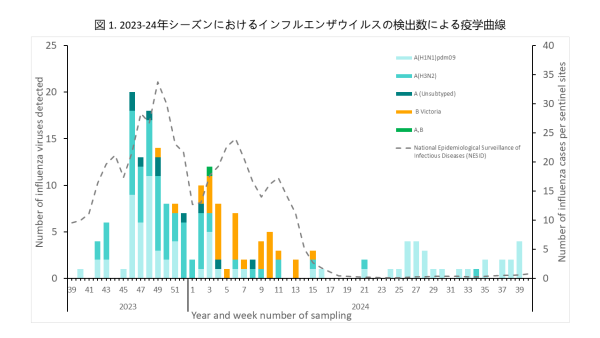
インフルエンザウイルスが検出された患者数を、検体採取日を基に週ごとに集計し、インフルエンザの疫学曲線を作成しました(図1)。インフルエンザウイルスは調査開始当初の2023年10月(第40週)から検出され、11月中旬(第46週)にピークに達しました。その後、患者数は徐々に減少し、本州では2024年4月頃(第15週)まで流行が続きました。新型コロナ感染症パンデミック以前は、1月から2月に流行のピークがみられましたが、2023年は流行の開始時期が早まりました。一方、沖縄では2024年4月以降もインフルエンザの患者発生が継続し、調査期間が終了する9月下旬まで、少数ながら毎月患者が確認されました。
インフルエンザの型・亜型別では、流行当初からA(H1N1)pdm09とA(H3N2)が同時に流行する状況が見られました。B型(B/Victoria系統)は12月に少数が検出されましたが、2024年1月下旬にピークを迎え、その後4月中旬まで流行の主体となりました(図1)。一方、2024年5月以降は、主に沖縄でインフルエンザが検出されていましたが、A(H1N1)pdm09に移行しました。
また、インフルエンザウイルスの薬剤耐性マーカーとなる変異については、バロキサビルおよびオセルタミビルに対する感受性低下を示す変異を調査しました。これらの変異は、RT-PCR、次世代シーケンシング(NGS)、およびサンガー法を用いて、PA遺伝子およびNA遺伝子における変異を確認しました。
治療前に採取された検体から検出されたインフルエンザウイルスは、A(H1N1)pdm09が80件、A(H3N2)が68件、B/Victoriaが33件(いずれも初診時患者株)でした。これら全てについて、RT-PCR法およびNGS法による解析の結果、バロキサビルに対する薬剤感受性低下を示す変異は検出されませんでした(表2)。
初診時に1回目の検体を採取し、その後、バロキサビルによる治療を受けた患者のうち、可能な患者については3~7日後の再受診時に2回目の検体(バロキサビル投与後検体)を採取しました。RT-PCR法により、インフルエンザA(H3N2)患者14名の検体を解析した結果、PA/I38T変異が1件で検出され、出現頻度は7.1%(1/14)でした(表2)。なお、バロキサビル投与後の検体がRT-PCRで陽性となった9例を分母とした場合、PA/I38T変異株の頻度は11.1%(1/9)に上昇しました。さらに、RT-PCR法でPA遺伝子変異が確認されたA(H3N2)検体についてNGS解析を行ったところ、同様にPA/I38T変異が確認されました。この検体に含まれるウイルスのうち、変異ウイルスが占める割合は87.9%と推定されました。
インフルエンザB/Victoria陽性で、バロキサビル投与後の4人の患者検体のうち1件からNGS法でPA/I38T 変異が確認されました(25.0%、1/4)。この検体に含まれるウイルスの中で、変異ウイルスが占める割合は、61.1%と推定されました。また、この検体は、検体中のウイルス量が少ないことから、RT-PCR法では変異の有無を確認できませんでした。インフルエンザウイルスB/VictoriaでI38T 変異が確認されたのは、本調査で初めての事例です。
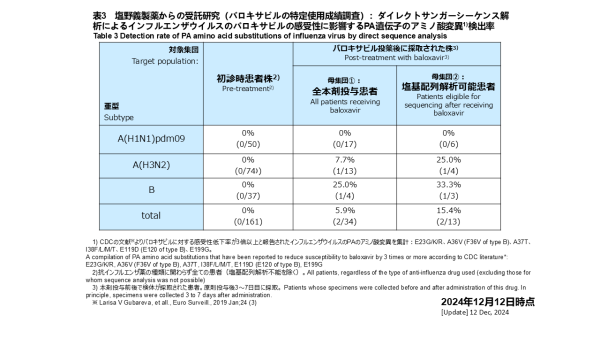
サンガー法によるダイレクトシーケンスによる解析では、現時点で解析が終了している検体のうち解析が可能であった初診時161検体のインフルエンザウイルスに対し、バロキサビルの感受性低下に関与するPA遺伝子の変異について解析しました(表3)。A(H1N1)pdm09、A(H3N2)、B型インフルエンザウイルスいずれにおいても、PA遺伝子の薬剤耐性変異は検出されませんでした。一方、バロキサビル治療をうけたA(H3N2)陽性患者13例のうち投薬後の検体1件にPA遺伝子の薬剤耐性変異が確認され、頻度は7.7%(1/13)でした。同じくB型陽性患者4例のうち投薬後の検体1件にPA遺伝子の薬剤耐性変異が確認され、頻度は25.0%(1/4)でした。また、バロキサビル投薬後の検体でサンガー法により解析できたインフルエンザA(H3N2)ウイルス4件、B型ウイルス3件を分母にすると、それぞれ頻度は25.0%(1/4)、33.3%(1/3)でした。
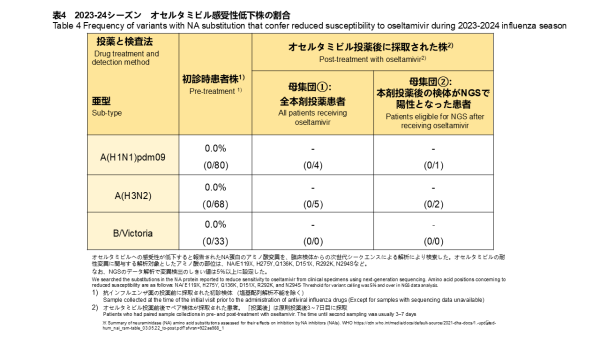
オセルタミビルの感受性に影響を与えるNA遺伝子の変異について、NGS解析を実施しました。治療前に採取された181検体すべてで、NA遺伝子にオセルタミビルに対する薬剤耐性変異は検出されませんでした(表4)。また、オセルタミビル治療後に採取された検体は、インフルエンザA(H1N1)pdm09陽性患者が4検体、A(H3N2)陽性患者が5検体でした。これら全ての再診時検体についても、オセルタミビル耐性変異は確認されませんでした(表3)。
Overview of Circulating Influenza Viruses and Antiviral Reduced-Susceptibility Strains in Japan during the 2023-2024 Season (as of December 25, 2024)
Between October 2023 and September 2024, we conducted a study across Japan to monitor influenza activity and assess the occurrence of antiviral reduced-susceptibility influenza strains against baloxavir and oseltamivir.
Eight outpatient medical facilities nationwide participated in the study. Upper respiratory tract swabs were collected from patients diagnosed with influenza-like illness (ILI) who tested positive using rapid diagnostic test kits (RDT). All clinical specimens were sent to Niigata University, where RT-PCR was used to detect the type and subtype of influenza viruses. Of the 209 collected specimens, 80 (38.3%) were A(H1N1)pdm09, 68 (32.5%) were A(H3N2), 10 (4.8%) were unsubtyped influenza A, 33 (15.8%) were B/Victoria, 1 (0.5%) showed coinfection with A and B viruses, and 17 (8.1%) were negative by RT-PCR (Table 1).
Influenza Epidemic Curve
The number of influenza virus-positive patients was aggregated weekly based on sample collection dates to construct an epidemic curve (Figure 1). Influenza viruses were detected from October 2023 (week 40) at the beginning of the study, with a peak in mid-November (week 46). Thereafter, case numbers gradually decreased, and in Honshu, influenza circulation persisted until approximately April 2024 (week 15). Before the COVID-19 pandemic, the seasonal peak typically occurred between January and February; however, in 2023, the season started earlier. Meanwhile, in Okinawa, influenza cases continued beyond April 2024, with sporadic cases observed monthly until the study concluded in late September.
Influenza Subtypes and Circulation Patterns
From the early stages of the season, both A(H1N1)pdm09 and A(H3N2) were co-circulating. B/Victoria was detected in small numbers in December, peaked in late January 2024, and became the dominant strain until mid-April (Figure 1). From May 2024 onward, influenza viruses were mainly detected in Okinawa, and the predominant virus shifted to A(H1N1)pdm09.
Investigation of Genetic Markers with Reduced Susceptibility to Antivirals.
We investigated genetic markers with reduced susceptibility to baloxavir and oseltamivir. Substitutions in the PA and NA genes associated with reduced susceptibility to these antivirals were examined using RT-PCR, next-generation sequencing (NGS), and Sanger sequencing.
Pre-Treatment Samples
From pre-treatment samples, 80 A(H1N1)pdm09, 68 A(H3N2), and 33 B/Victoria (all initial consultation samples) were analyzed. No substitutions associated with reduced susceptibility to baloxavir were detected by RT-PCR or NGS (Table 2).
Post-Baloxavir Treatment Samples
Initial samples were collected at the first visit, and post-treatment samples were collected 3–7 days later for patients who returned for follow-up after baloxavir treatment. Among 14 A(H3N2) patient samples, one exhibited the PA/I38T substitution, with a prevalence of 7.1% (1/14) (Table 2). When restricted to nine RT-PCR-positive post-treatment samples, the prevalence increased to 11.1% (1/9). NGS confirmed the PA/I38T substitution in the A(H3N2) sample, estimating that mutant viruses constituted 87.9% of the viral population in the sample.
One of the B/Victoria-positive post-treatment samples from four patients exhibited the PA/I38T substitution via NGS (25.0%, 1/4), with mutant viruses accounting for 61.1% of the viral population. Due to the low viral load, RT-PCR could not detect PA/I38T substitution. This was the first detection of the I38T substitution in B/Victoria in this study.
Sanger Sequencing Analysis
Among 161 analyzable initial consultation samples, no substitutions associated with baloxavir resistance in the PA gene were detected for A(H1N1)pdm09, A(H3N2), or B viruses (Table 3). However, one post-treatment sample from 13 A(H3N2)-positive patients exhibited PA gene substitutions, with a prevalence of 7.7% (1/13). Similarly, one post-treatment sample from four B-positive patients exhibited PA gene substitutions, with a prevalence of 25.0% (1/4). When restricted to Sanger-analyzable post-treatment samples, frequencies were 25.0% (1/4) for A(H3N2) and 33.3% (1/3) for B viruses.
Oseltamivir Resistance
NGS analysis of the NA gene was performed to assess oseltamivir resistance. No oseltamivir-resistant NA gene substitutions were detected in all 181 pre-treatment samples (Table 4). Post-treatment samples included four from A(H1N1)pdm09-positive patients and five from A(H3N2)-positive patients. No resistance substitutions were detected in these samples (Table 3).