2022-2023年シーズンの日本におけるインフルエンザサーベイランス(2024年10月17日更新)
2022-2023年シーズンの日本におけるインフルエンザ流行株と抗インフルエンザ薬感受性低下株調査の概要(2024年10月17日更新)
2022年10月から2023年9月までの期間中、日本各地でインフルエンザの流行動向とバロキサビルおよびオセルタミビルに対する薬剤耐性変異を持つインフルエンザウイルスの発生状況について調べました。
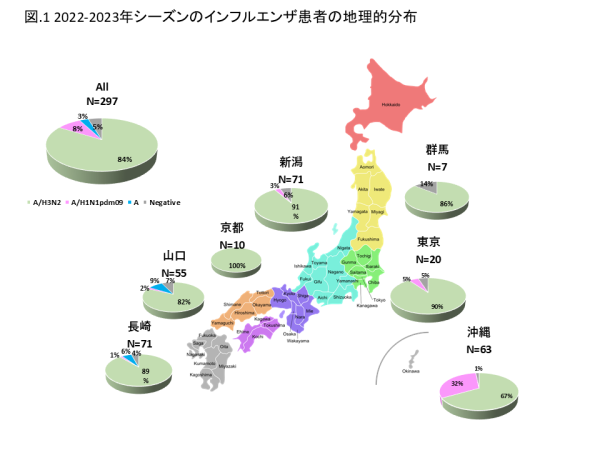
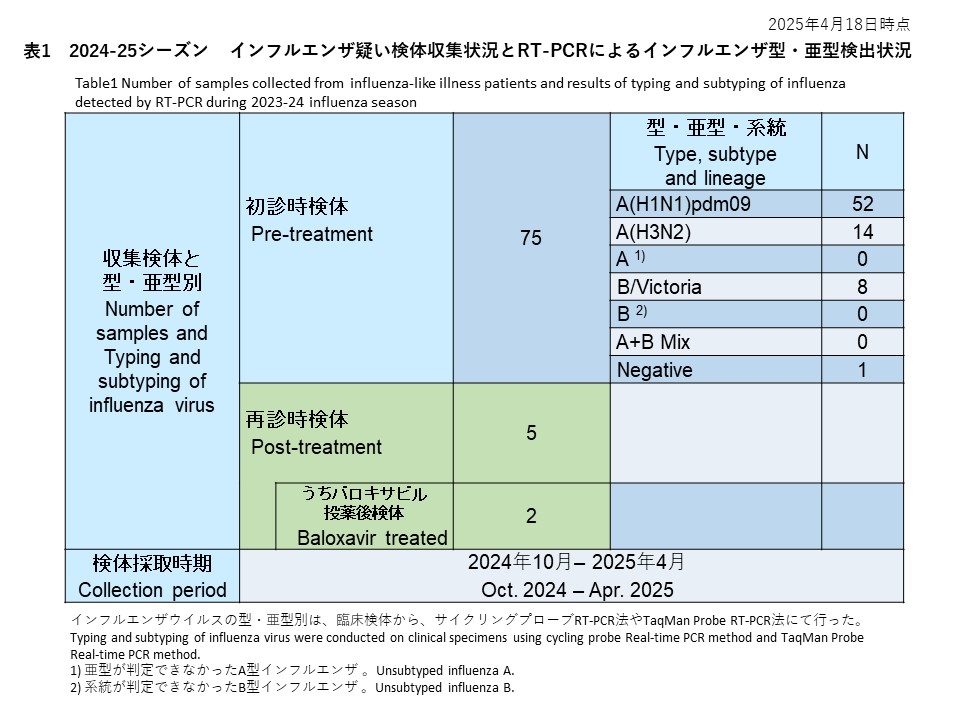
全国7県の8つの外来医療機関が研究に参加しました(図1)。医療機関を受診し、迅速診断テストキット(RDT)で陽性と判定されたインフルエンザ様疾患(ILI)患者から、上気道スワブを検体として収集しました。これらの臨床検体は全て新潟大学に送られ、RT-PCR法でインフルエンザウイルスの検出と型/亜型の決定を行いました。採取された297件の検体のうち、248件(83.5%)がインフルエンザウイルスA(H3N2)、25件(8.4%)がインフルエンザウイルスA(H1N1)pdm09、10件(3.4%)が亜型判定不能のインフルエンザウイルスA型、14件がRT-PCR法で陰性でした(図1、表1)。
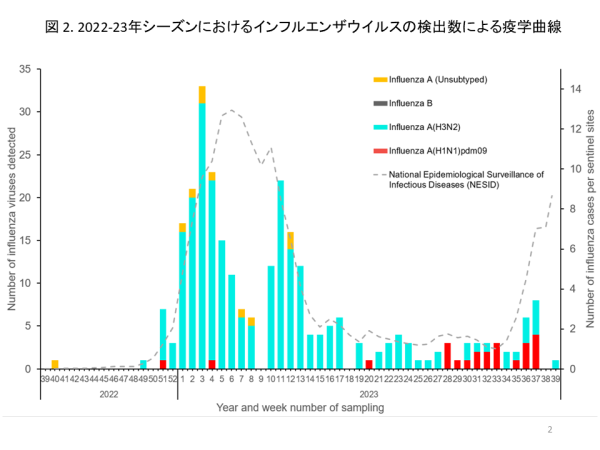
インフルエンザウイルスが検出された患者数を検体採取した日をもとに週毎に集計し、インフルエンザの疫学曲線を作成しました。インフルエンザの流行は2022年末に始まり、2023年1月にピークに達し、3月に小規模なピークが再度発生し、インフルエンザの流行は3月から9月まで低いレベルで続きました(図2)。日本各地で主にA(H3N2)のインフルエンザが流行しましたが、A(H1N1)pdm09のインフルエンザウイルスも新潟、東京、山口、沖縄で少数検出されました(図1、2)。
薬剤耐性マーカーとなる変異(バロキサビルに対する感受性低下変異のPA/I38T変異、およびオセルタミビルに対する耐性変異のNA/H275Y)についてRT-PCR法で検出し、続いて次世代シーケンシング(NGS)およびサンガー法によりPA遺伝子とNA遺伝子にみられる薬剤感受性低下変異を確認しました。初診時に1回目の検体を採取し、さらにバロキサビル又はオセルタミビル投薬治療後、3-7日後に再受診した際に2回目の検体を採取しました。
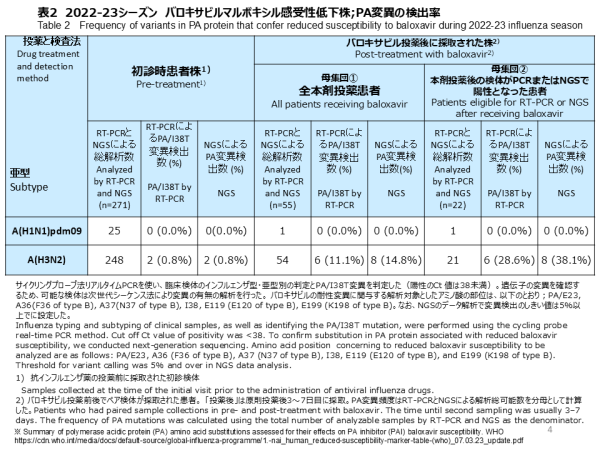
RT-PCR法によりバロキサビルに対し薬剤感受性低下を示すPA/I38T変異が、初診時に採取された248件のインフルエンザA(H3N2)のうち2件で検出され、その頻度は0.8%(2/248)でした(表2)。NGSによりPA遺伝子変異の確認を行ったところ、RT-PCR法と同様に2件からPA/I38T変異を検出しました。しかしながら、その他のPA変異株は確認できませんでした(0.8%、2/248)。なお、初診時に採取された25件のインフルエンザA(H1N1)pdm09から、PA変異株は、RT-PCR法とNGS共に検出されませんでした(表2)。
バロキサビル投薬治療後の再診時の患者検体におけるRT-PCR法によるPA/I38T変異の検出では、インフルエンザA(H3N2)陽性患者54名のうち6名から検出されたため、出現頻度は11.1%(6/54)となりました(表2)。ちなみに投薬後再診時の検体がRT-PCR法で陽性となった21例を分母とした場合には、PA/I38T変異株の頻度は28.6%(6/21)に上昇しました。同じ検体で、NGS解析を行ったところ、薬剤耐性に関係するPA変異が投薬後の検体から8件見つかりました(14.8%、8/54)(表2)。その変異の内訳は、PA/I38T変異が4件、PA/I38M変異が1件、PA/I38M/TおよびPA/E23K+I38T、PA/E23G+I38Tの混合変異がそれぞれ1件ずつでした。バロキサビル投薬後に採取されNGSまたはRT-PCR法による解析が可能であった21検体を母数とすると、頻度は38.1%(8/21)となりました。バロキサビルで治療されたインフルエンザA(H1N1)pdm09患者1名の投薬後検体に、PA変異はRT-PCR法、NGS共に検出されませんでした (0/1) (表2)。
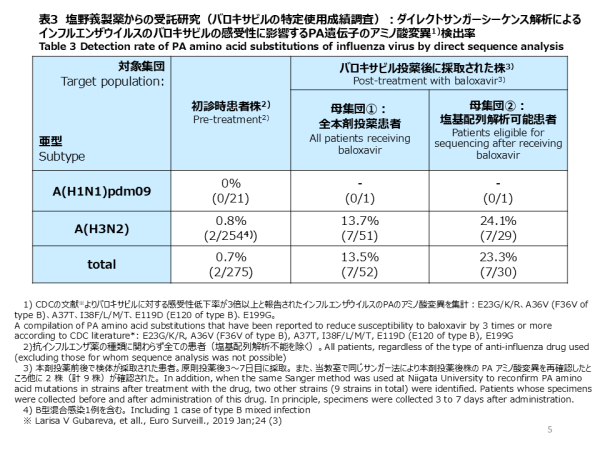
サンガー法によるダイレクトシーケンス解析が可能であった275検体のインフルエンザウイルスに対し、バロキサビルの感受性低下に関与するPA遺伝子の変異について解析しました(表3)。治療前の初診患者から検出されたA(H1N1)pdm09インフルエンザウイルス21件には、PA遺伝子の薬剤耐性変異は検出されませんでした(0/21)。一方、A(H3N2)インフルエンザウイルス254件中2件でPA/I38T変異が初診時の患者から確認され、頻度は0.8%(2/254)でした。バロキサビル投薬前と投薬後の2回検体が採取されたA(H1N1)pdm09陽性患者1例の投薬後に採取された検体には、PA遺伝子の薬剤耐性変異は検出されませんでした。バロキサビル治療をうけたA(H3N2)陽性患者51例のうち投薬後の検体7件にPA遺伝子の薬剤耐性変異が確認され、頻度は13.7%(7/51)でした。また、バロキサビル投薬後の検体でサンガー法により解析できたインフルエンザA(H3N2)ウイルス29件を分母にすると、頻度は24.1%(7/29)に上昇しました。
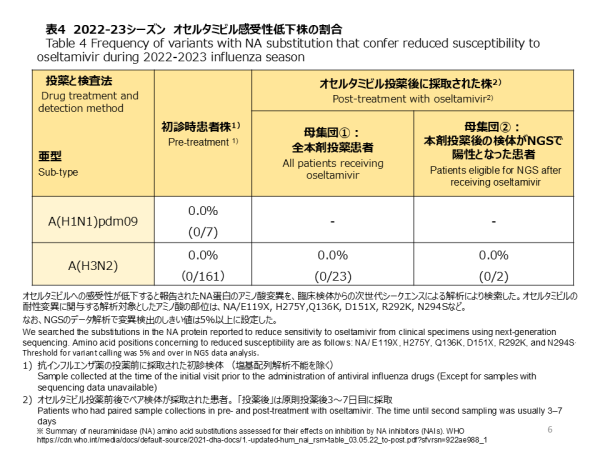
オセルタミビルの感受性に影響するNA遺伝子の変異はNGSで解析しました。治療前初診時に採取された7例の検体から検出されたA(H1N1)pdm09インフルエンザウイルス、及び161例のA(H3N2) ウイルスには、NA遺伝子にオセルタミビルに対する薬剤耐性変異は見つかりませんでした(表4)。オセルタミビル投薬前と投薬後の2回検体が採取されたA(H3N2)陽性患者23例については、投薬後の再診時の検体からオセルタミビル耐性変異は確認されませんでした(0/23)。同様に、投薬後再診時に採取されNGSによる解析が可能であった2件のA(H3N2)を分母とした場合にも、NA遺伝子の薬剤耐性変異はなかったため頻度は0.0%でした(0/2)(表4)。
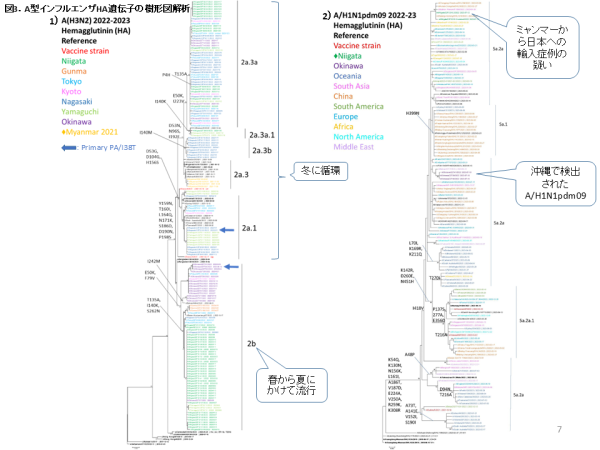
A型インフルエンザウイルスについて、HA遺伝子の系統樹解析を行いました。2022-23年シーズンに日本で流行したA(H3N2)インフルエンザウイルスは複数の遺伝的クレードに分かれており、冬季に流行したウイルスは、ほとんどクレード2a.3aに属し、春から夏にかけて流行したウイルスはクレード2bに属しました(図3-1)。初診時の検体から検出された2件のPA/I38変異株は、それぞれ2a.1と、2bという別のHAクレードに属していました。これはPA変異株が1つの系統からクローナルな感染伝播を起こしている状態では無いことを示してます。一方、7月初旬に沖縄で採取されたA(H1N1)pdm09インフルエンザウイルスは5a.2aに属し、中東、東京、欧州の株と遺伝的に類似していました。ミャンマーから7月に日本に輸入されたと考えられるA(H1N1)pdm09ウイルスは、5a.2aに属しており、中国やタイで検出された株に近似していました(図3-2)。
Summary results of influenza virus detection and variant strains that confer reduced susceptibility to antivirals in Japan in 2022-2023 season. Updated on 2024.10.17.
From October 2022 to September 2023, we monitored influenza virus circulation and variant strains that confer reduced susceptibility to Baloxavir or Oseltamivir in Japan.
Eight outpatient clinics from seven prefectures nationwide participated in the study (Figure 1). Upper respiratory swabs were collected from influenza-like illness (ILI) patients who visited any of the collaborative clinics and tested positive by rapid diagnostic test kit (RDT). After transportation to Niigata University, all clinical samples were examined by RT-PCR for typing/subtyping of influenza virus. Among 297 received clinical samples, 248 (83.5%) were identified as A(H3N2), 25 (8.4%) as A(H1N1)pdm09, 10 (3.4%) as unsubtyped influenza A, and 14 were negative for influenza virus by RT-PCR (Table 1). Influenza A(H3N2) predominantly circulated throughout Japan, although a few A(H1N1)pdm09 viruses were detected in Niigata, Tokyo, Yamaguchi, and Okinawa (Figure 1, Table 1).
Influenza activity started in late 2022 and peaked in January (Figure 2). A secondary small peak was detected in March 2023, which gradually decreased. Influenza circulation continued at a low level from March to September 2023 (Figure 2). Influenza A(H3N2) predominantly circulated throughout the studied areas, but A(H1N1)pdm09 viruses were also detected in Niigata, Tokyo, Yamaguchi, and Okinawa (Figures 1 and 2).
Amino acid substitutions in the influenza virus genome as drug-resistant markers, PA/I38T in the PA gene for Baloxavir and NA/H275Y in the NA gene for Oseltamivir, were determined by RT-PCR. Subsequently PA or NA variants in relevant genes, which affect Baloxavir or Oseltamivir sensitivity were confirmed by next-generation sequencing (NGS) and the Sanger method. A first sample was collected at the initial visit (pre-treatment), and a second sample was collected at a follow-up visit 3-7 days after treatment with baloxavir or oseltamivir.
By RT-PCR, the substitution with reduced susceptibility to Baloxavir (PA/I38T) was observed in two out of 248 A(H3N2) viruses before treatment, representing a frequency of 0.8% (2/248) (Table 2). Using NGS to verify PA gene substitutions, we detected the PA/I38T mutation in 2 cases, consistent with RT-PCR results. However, no other PA variants were detected (0.8%, 2/248). On the contrary, no PA variants were detected in the 25 pre-treatment influenza A(H1N1)pdm09 samples using both RT-PCR and NGS methods (Table 2).
Among A(H3N2) patients treated with Baloxavir (n=54), six cases with PA/I38T substitution were detected from clinical samples at the second visit by RT-PCR, defining the frequency of 11.1% for PA/I38T based on the number of Baloxavir-treated patients (6/54) (Table 2). If the denominator is based on 21 cases positive by RT-PCR at the second visit, the frequency rose to 28.6% (6/21). We conducted NGS analysis on paired samples from the 54 A(H3N2) patients. Eight cases with PA substitutions were found after baloxavir treatment, representing a frequency of 14.8% (8/54). Of these eight cases, four were PA/I38T, one was PA/I38M, and one each of mixed substitutions PA/I38M/T, PA/E23K+I38T, and PA/E23G+I38T. When we use the denominator based on 21 cases positive by RT-PCR and eligible for NGS at the second visit after Baloxavir treatment, the frequency became 38.1% (8/21) (Table 2). No PA variants (0/1) were detected by both RT-PCR and NGS after treatment with Baloxavir in one patient with influenza A(H1N1)pdm09 (Table 2).
Using Sanger method sequencing, 275 pre-treatment viruses were analyzed (Table 3). Among influenza A(H1N1)pdm09 strains sequenced before treatment (n=21), no PA substitutions were found (0/21). Out of 254 pre-treated influenza A(H3N2) viruses sequenced, two were found to harbor the PA/I38T substitution, with a frequency of 0.8% (2/254). In A(H1N1)pdm09, no PA variant was detected in one patient who had the second sampling after Baloxavir treatment (0/1) (Table 3). Among 51 A(H3N2) who had the second sampling after Baloxavir treatment, seven possessed PA variants, representing a frequency of 13.7% (7/51). Based on 29 cases eligible to be sequenced by the Sanger method at the second visit, the frequency rose to 24.1% (7/29).
We analyzed NA gene by NGS to find NA variants that affect sensitivity to Oseltamivir. NGS results showed no substitution in the NA gene affecting sensitivity to Oseltamivir in seven A(H1N1)pdm09 cases and 161 A(H3N2) cases before treatment (Table 4). Among 23 Oseltamivir-treated cases sampled twice at the first and second visits, no NA variants were detected by NGS analysis in the second visit samples (0/23). If we base the denominator on the two samples successfully analyzed by NGS at the second visit after Oseltamivir treatment, the frequency remained 0.0% (0/2) (Table 4).
We performed a phylogenetic analysis of the HA gene of influenza A viruses. Influenza A(H3N2) viruses circulated in Japan had several genetic clades; however, most collected viruses split into two clades (2a.3a and 2b). The clade 2a.3a consists mainly of viruses circulated in winter, and the 2b clade was formed from spring-summer strains (Figure 3-1). Two viruses harboring the PA/I38T substitution collected at the pre-treatment visit, marked with blue arrows, belonged to different HA clades: one to 2a.1 and the other to 2b (Figure 3-1). This indicates no evidence for a clonal spread of the primary PA variants in the community. Influenza A(H1N1)pdm09, collected in early July in Okinawa, belonged to clade 5a.2a and had close genetic similarity with strains from the Middle East, Tokyo, and Europe (Figure 3-2). However, one A(H1N1)pdm09 virus, suspected to be imported from Myanmar to Japan in July, belonged to clade 5a.2a and had genetic proximity to strains from China and Thailand.