1.研究概要
近年、ゲノム情報の解明を背景に、網羅的解析が盛んに行われ、様々な生命現象や疾患をシステムとして理解する試みがなされています。中でも生命現象と直接的に結び付くタンパク質の変化をグローバルにとらえるプロテオーム解析の重要性は明白です。システム生化学分野では様々な生命現象の理解や各種疾患の原因解明および診断法開発に向けて、生命現象の直接的な担い手であるタンパク質の存在量を大規模かつ正確に測定する技術の開発とそれを“がん”をはじめとする各種疾患の分子基盤の解明を目指しています。
詳しい内容については、システム生化学分野ホームページをご参照ください。
2.研究内容
- ・質量分析を基盤とするプロテオーム解析技術の開発
- ・タンパク質の量や機能の精密計測に基づくシステム生物学
- ・先進プロテオミクス用いた各種疾患発症機構や分子病態の解明
3.研究の成果
[分野] システム生化学
[研究テーマ] タンパク質精密絶対定量技術の開発
[内容]
近年の質量分析計の著しい高性能化と種々の定量技術の開発によってプロテオーム解析技術は成熟し、一度に数千のタンパク質の同定や定量が可能となったことで、様々な生物学領域の発展に大いに貢献している。その一方で、現在普及している手法では定量性や再現性の問題に直面しており、新たな方法論の構築が望まれてきた。近年、従来のプロテオーム解析法の問題点を解消できる手法として、MRM (multiple-reaction monitoring) などの技術を用いたターゲット・プロテオミクスが確立され、任意のタンパク質のハイスループットかつ正確な定量が可能となった。しかしながら、MRMを実施するために必要な情報リソースの不足とそのメソッド構築の煩雑さから、広く普及するに至っていない。そこで、質量分析計によるMRM 法とヒト組み換えタンパク質リソース(約20,000遺伝子) とを組み合わせることで、これまで不可能とされてきた全タンパク質の絶対定量を可能とする画期的なプロテオーム解析法(iMPAQT法)を開発した。
[写真など]
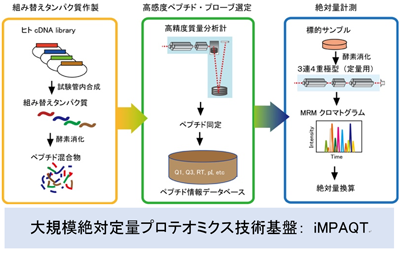
[分野] システム生化学
[研究テーマ] 機能プロテオーム解析によるがん研究
[内容]
iMPAQT法を用いた代謝酵素の網羅的絶対定量によって、細胞がん化における代謝システムの変遷を定量的に明らかにした。また、タンパク質リン酸化やユビキチン化、さらにはタンパク質相互作用解析法を駆使することで、がん特有の分子ネットワークの解明やそれを標的とした創薬を目指している。
[写真など]
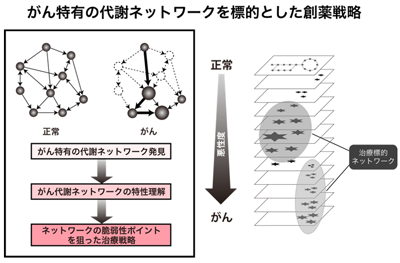
[参考文献]
1) Matsumoto M, Nakayama KI. The promise of targeted proteomics for quantitative network biology. Curr. Opin. Biotechnol. 54: 88-97, 2018.
2) Nakatsumi H, Matsumoto M, Nakayama KI. Noncanonical pathway for regulation of CCL2 expression by an mTORC1-FOXK1 axis promotes recruitment of tumor-associated macrophages. Cell Rep. 21: 2471-2486, 2017.
3) Matsumoto M, Matsuzaki F, Oshikawa K, Goshima N, Mori M, Kawamura Y, Ogawa K, Fukuda E, Nakatsumi H, Natsume T, Fukui K, Horimoto K, Nagashima T, Funayama R, Nakayama K, Nakayama KI. A large-scale targeted proteomics assay resource based on an in vitro human proteome. Nat. Methods 14: 251-258, 2017.
4) Matsumoto A, Pasut A, Matsumoto M, Yamashita R, Fung J, Monteleone E, Saghatelian A, Nakayama KI, Clohessy JG, Pandolfi PP. mTORC1 and muscle regeneration are regulated by the LINC00961-encoded SPAR polypeptide. Nature 541: 228-232, 2017.
5) Yumimoto K, Matsumoto M, Oyamada K, Moroishi T, Nakayama KI. Comprehensive identification of substrates for F-box proteins by differential proteomics analysis. J. Proteome Res. 11: 3175-85, 2012.
6) Oshikawa K, Matsumoto M, Oyamada K, Nakayama KI. Proteome-wide identification of ubiquitylation sites by conjugation of engineered lysine-less ubiquitin. J. Proteome Res. 11: 796-807, 2012.
7) Matsumoto M, Oyamada K, Takahashi H, Sato T, Hatakeyama S, Nakayama KI. Large-scale proteomic analysis of tyrosine-phosphorylation induced by T-cell receptor or B-cell receptor activation reveals new signaling pathways. Proteomics 9: 3549-63, 2009.
8) Matsumoto M, Hatakeyama S, Oyamada K, Oda Y, Nishimura T, Nakayama KI. Large-scale analysis of the human ubiquitin-related proteome. Proteomics 5: 4145-51, 2005.
9) Matsumoto M, Yada M, Hatakeyama S, Ishimoto H, Tanimura T, Tsuji S, Kakizuka A, Kitagawa M, Nakayama KI. Molecular clearance of ataxin-3 is regulated by a mammalian E4. EMBO J. 23: 659-69, 2004.






