1.研究概要
ミトコンドリアは、細胞が消費するATPの大部分を産生する細胞の活動に必須のオルガネラであり、ミトコンドリア異常は様々な病気の原因となる。このため、細胞にはミトコンドリアを正常に保つために機構が幾つか備わっており、そのうちの一つがオートファジーによるミトコンドリア分解(ミトコンドリアオートファジーまたはmitophagy)である。我々の研究は、mitophagyの分子機構から生理的意義まで、様々な生物種を用いて解明し、ミトコンドリア関連疾患の病態解明と治療法開発に応用することを目指している。
詳しい研究内容については、機能制御学ホームページをご覧ください。
2.研究テーマ
- (1)マウスモデルや培養細胞を用いて、mitophagyと疾患との関係を解明する
- (2)mitophagyを利用したミトコンドリア関連疾患に対する創薬
- (3)出芽酵母や分裂酵母などのモデル生物を用いたmitophagyの分子機構解明
3.研究の成果
[分野] 機能制御学
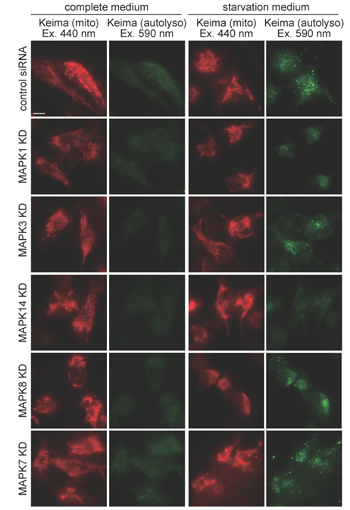
[研究テーマ] 哺乳類でのmitophagy観察法とその応用
[内容]
KeimaはpH依存性の蛍光タンパク質で、中性環境では440nmで酸性環境では560nmで励起されるという特徴を持っている。このため、ミトコンドリア移行シグナルを付けたmito-Keimaはミトコンドリアに局在するが、mitophagyでリソソームに運ばれた時のみ、リソソーム内が酸性のため560nmで励起されるようになる。この蛍光を検出することによりmitophagyを観察する。この方法を用いて、様々なMAPキナーゼとmitophagyの関係を調べたところ、p38やErk2のシグナル経路がmitophagyに重要な役割を持っていることを明らかにした。
[参考文献]
Yamashita SI, Jin X, Furukawa K, Hamasaki M, Nezu A, Otera H, Saigusa T, Yoshimori T, Sakai Y, Mihara K, Kanki T. Mitochondrial division occurs concurrently with autophagosome formation but independently of Drp1 during mitophagy. J Cell Biol. 2016;215(5):649-665
Hirota Y, Yamashita S, Kurihara Y, Jin X, Aihara M, Saigusa T, Kang D, Kanki T. Mitophagy is primarily due to alternative autophagy and requires MAPK1 and MAPK14 signaling pathways. Autophagy. 2015;11(2):332-343.
[写真など]
[分野] 機能制御学
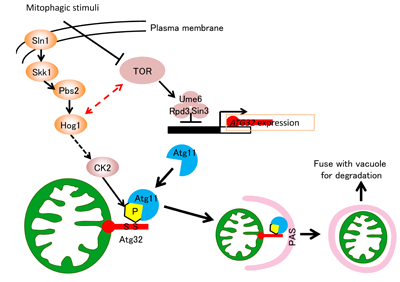
[研究テーマ] mitophagyの分子機構
[内容]
まず出芽酵母でマイトファジーを観察する方法を確立し、細胞質に存在するAtg11がmitophagyに必須であることを見いだした。(Kanki
& Klionsky, JBC 2008)。次に、mitophagy関連因子のスクリーニングを行い(Kanki et al. MBC 2009)、最終的にミトコンドリア分解の目印となるミトコンドリア外膜タンパクAtg32を同定することに成功した(Kanki
et al. Dev. Cell 2009)。Atg32の解析を進め、図に示すようにミトコンドリア上の目印であるAtg32が、細胞質のAtg11に識別され捕まえられる(結合する)ことによって、選択的なミトコンドリア分解が開始することを明らかにした。さらに、Atg32の114番目のセリン残基のリン酸化がAtg32とAtg11の結合に必須であること(Aoki
et al. MBC 2011)、Atg32のリン酸化はCasein Kinase 2が行っていること(Kanki et al. EMBO Rep.
2013)や、マイトファジーの制御、即ちAtg32のリン酸化にはHog1 MAPキナーゼシグナル経路が重要であること(Aoki et al.
MBC 2011)、TORの下流でUme6-Sin3-Rpd3複合体がAtg32遺伝子発現を抑制していること(Aihara et al. JCS
2014)などを出芽酵母におけるマイトファジーの分子機構の多くの解明に成功している(図参照)。
[写真など]
[参考文献]
Aihara M, Jin X, Kurihara Y, Yoshida Y, Matsushia Y, Oku M, Hirota Y, Saigusa
T, Aoki Y, Uchiumi T, Yamamoto T, Sakai Y, Kang D, *Kanki T.
Tor and the Sin3-Rpd3 complex regulate expression of the mitophagy receptor
protein Atg32 in yeast.
J Cell Sci. 2014;127:3184-96.
Kanki T, Kurihara Y, Jin X, Goda T, Ono Y, Aihara M, Hirota Y, Saigusa
T, Aoki Y, Uchiumi T, Kang D.
Casein kinase 2 is essential for mitophagy.
EMBO Rep. 2013: 14(9): 788-94.
Kurihara Y, *Kanki T, Aoki Y, Hirota Y, Saigusa T, Uchiumi T, Kang D.
Mitophagy plays an essential role in reducing mitochondrial production
of reactive oxygen species and mutation of mitochondrial DNA by maintaining
mitochondrial quantity and quality in yeast.
J Biol Chem. 2012;287(5):3265-72.
Aoki Y, Kanki T, Hirota Y, Kurihara Y, Saigusa T, Uchiumi T, Kang D.
Phosphorylation of Ser114 on Atg32 mediates mitophagy.
Mol Biol Cell. 2011;22(17):3206-17.
Kanki T, Wang K, Baba M, Bartholomew CR, Lynch-Day MA, Du Z, Geng J, Mao
K, Yang Z, Yen WL, Klionsky DJ.
A genomic screen for yeast mutants defective in selective mitochondria
autophagy.
Mol Biol Cell. 2009;20(22):4730-8.
Kanki T, Wang K, Cao Y, Baba M, Klionsky DJ.
Atg32 is a mitochondrial protein that confers selectivity during mitophagy.
Dev Cell. 2009;17(1):98-109.
Kanki T, Klionsky DJ.
Mitophagy in yeast occurs through a selective mechanism.
J Biol Chem. 2008;283(47):32386-93.






