抗酸菌における決定的な休眠誘導機構を発見 − 天然変性タンパク質による新規のDNA凝集メカニズム −
新潟大学大学院医歯学総合研究科細菌学分野の西山晃史講師、松本壮吉教授らの研究グループは、京都大学複合原子力科学研究所(清水将裕助教)、金沢大学ナノ生命科学研究所(古寺哲幸教授ら)、九州大学生体防御医学研究所(眞胗浩太講師[現・同大学大学院薬学研究院])、大阪公立大学(山口雄大助教[現・国立感染症研究所])、新潟大学大学院自然科学研究科(伊東孝祐准教授)との共同研究で、抗酸菌の天然変性ヒストン様タンパク質mycobacterial DNA-binding protein 1(MDP1)が、他の核酸結合タンパク質では報告のない、天然変性領域*1を介した新規のDNA凝集メカニズムを介して抗酸菌の休眠を誘導することを明らかにしました。
【本研究成果のポイント】
・結核や非結核性抗酸菌(NTM)症などの抗酸菌症は、難治性であり、投薬期間は半年から年単位に及ぶ。この根底に、休眠して薬剤パーシスター*2化する抗酸菌の性質があるが、休眠期に誘導される分子の解析が盛んに行われてきた中で、実際に休眠を誘導する分子は報告されていなかった。
・結核は人類史上最も多くの人命を奪ってきた。2022年の死亡者数は157万人に上った。休眠結核菌による潜伏感染(潜在性結核*3)は世界人口の1/4に及ぶとされ、活動性結核の主要な発生源である。休眠菌は多くの薬剤にも抵抗性で、長期投薬を要する元凶である。
・抗酸菌のヒストン様タンパク質は、細菌では希なヒストンに似た天然変性領域を持ち、その作用で休眠を誘導する。
・本研究グループでは、高速原子間力顕微鏡(高速AFM)*4によるタイムラプス撮影と、分子動力学計算を組み合わせることによって、MDP1の天然変性領域が両面テープのように2本のDNA二重らせんを貼り合わせていくという、他の核酸結合タンパク質では報告されていない、新規のDNA凝集メカニズムを解明した。
・MDP1の天然変性領域は、抗酸菌と一部のストレス耐性菌に特徴的で、強力な休眠誘導活性を持っていた。本研究成果は、未知であった抗酸菌の休眠誘導の決定的なメカニズムを明らかにした。また本成果は、休眠菌にも有効で、既存薬との交差耐性が無い薬剤の開発にも繋がることが期待される。
Ⅰ.研究の背景
細胞は増殖を停止すると老化し、次第に死に近づきますが、一部の細菌は、増殖を停止した後も長期間、生き続けることが知られています。これを休眠「dormancy」と呼びます。休眠すると薬剤にも擬似的に耐性(Tolerance)となることから、病原性細菌が休眠すると、難治性となるケースが多いです。その好例が、結核や非結核性抗酸菌(NTM)症などの抗酸菌による感染症です。
結核は人類史上最も多くの人命を奪ってきた感染症であり(Paulson, Nature 2013)、2022年の死亡者数は157万人に上りました(HIV陽性患者を含む, WHO, World Tuberculosis Report 2023)。結核では、休眠結核菌による潜伏感染(潜在性結核)が世界人口の1/4に及ぶと推計され、活動性結核の主要な発生源となっています。また、休眠菌は多くの薬剤に抵抗性であり、結核の治療に長期投薬を要する元凶となっています。
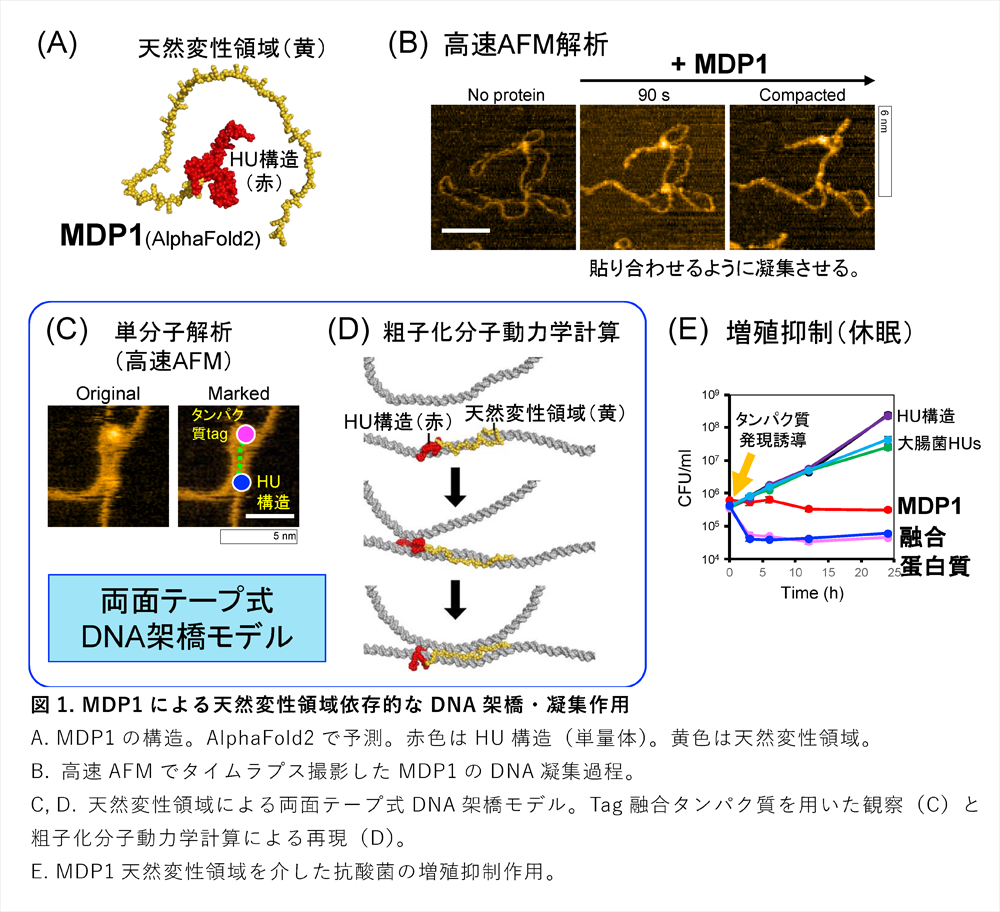
結核菌を含めた全ての抗酸菌が発現するMDP1は、全ての細菌が発現するヒストン様タンパク質HUの仲間です。興味深いことに、MDP1のHU構造には、細菌では非常に希なヒストンに似た天然変性領域が付加されています(図1A)。MDP1は低酸素休眠菌で高発現します。天然変性領域の作用で休眠(染色体凝集、増殖抑制等)を誘導します。
Ⅱ.研究の概要
本研究では、MDP1の休眠誘導作用に関わる、天然変性領域を介したDNA凝集メカニズムの解明を目指しました。一定の構造を取らない天然変性領域は通常の構造解析が困難なため、分子の動きをナノメータースケールでタイムラプス撮影可能な高速AFMを用いて、MDP1によるin vitroでのDNA凝集過程を撮影しました。また、粗子化分子動力学計算*5による本DNA凝集過程の再現を組み合わせることによって、MDP1天然変性領域によるDNA架橋メカニズムを解明しました。
Ⅲ.研究の成果
MDP1が隣接する2本のDNA二重らせんを、ジッパーを閉じるように貼り合わせて束ねていく凝集過程を高速AFMによるタイムラプス撮影で捉え(図1B)、天然変性領域がこの活性を担っていることを明らかにしました。また、高速AFM解析で、天然変性領域の先端にtagを付加した融合タンパク質を用いることによって、DNA凝集過程の天然変性領域の動きを追跡することに成功しました(図1C)。さらに、粗子化分子動力学計算によって、MDP1のDNA凝集過程の計算モデルを構築しました(図1D)。これらの結果を組み合わせることによって、他の核酸結合タンパク質では報告がない、天然変性領域が2本のDNA二重らせんの間に伸長して静電引力で両面テープのように貼り合わせる新規のDNA架橋メカニズムを解明しました。このDNA架橋が連続的に形成されることにより、広範囲のDNA凝集を誘導すると考えられます。また、天然変性領域によるDNA架橋は可逆的な構造でした。
MDP1を抗酸菌で過剰発現させると、天然変性領域の作用で染色体凝集を誘導します。また、生存率を大きく低下させることなく増殖を抑制し、菌を休眠させます(図1E)。この天然変性領域を大腸菌HUに融合させると、大腸菌HUの活性を支配し、MDP1様のDNA凝集活性、強力な菌の休眠誘導活性を示しました(図1E)。
MDP1の天然変性領域は、抗酸菌と一部のストレス耐性菌に特徴的で、強力な休眠誘導活性を持っていました。本研究で解明したMDP1天然変性領域による可逆的なDNA架橋・凝集の構造モデルは、抗酸菌の休眠と覚醒の制御に適したメカニズムであることが示唆されました。
Ⅳ.今後の展開
休眠して、薬剤パーシスター化する抗酸菌の感染症は、治療期間が長く、難治性であることが特徴で、社会問題となっています。抗酸菌の休眠における重要なメカニズムが分かったことで、結核や非結核性抗酸菌(NTM)症などの抗酸菌症の創薬が大きく前進することが期待されます。
また、増殖を停止した後にも生き続ける「休眠」のメカニズムを解明していくことで、生物共通の長寿のメカニズムにも迫る可能性があります。
Ⅴ.研究成果の公表
本研究成果は、2024年1月25日発刊のオックスフォード大学出版の科学誌「Nucleic Acids Research」に掲載されました。
論文タイトル:Dynamic action of an intrinsically disordered protein in DNA compaction that induces mycobacterial dormancy(Volume 52, Issue 2, Pages 816-830)
著者:Akihito Nishiyama, Masahiro Shimizu, Tomoyuki Narita, Noriyuki Kodera, Yuriko Ozeki, Akira Yokoyama, Kouta Mayanagi, Takehiro Yamaguchi, Mariko Hakamata, Amina Kaboso Shaban, Yoshitaka Tateishi, Kosuke Ito, Sohkichi Matsumoto
doi: 10.1093/nar/gkad1149
Ⅵ.謝辞
本研究は、文部科学省(MEXT)日本学術振興会(JSPS)基盤研究B(20H03483)、基盤研究C(17K08823)、研究活動スタート支援(20K22629)、日本医療研究開発機構(AMED)新興・再興感染症に対する革新的医薬品等開発推進研究事業(20fk0108090、20fk0108089)、革新的先端研究開発支援事業AMED-CREST(22gm1610009)、科学技術振興機構(JST)戦略的創造研究推進事業CREST(JPMJCR1762)の助成、並びに新潟大学U-goグラント、世界トップレベル研究拠点プログラム(WPI、金沢大学ナノ生命科学研究所)、九州大学生体防御医学研究所(多階層生体防御システム研究拠点)による支援を受けて実施されました。
【用語解説】
*1 天然変性領域:一部のタンパク質の内部(または全体)に存在する領域。アミノ酸配列の低複雑性や偏ったアミノ酸組成のために一定の立体構造をとることができず、まるで変性しているかのように振る舞う。近年、様々な細胞機能(クロマチン制御等)、疾患(アルツハイマー病、筋萎縮性側索硬化症(ALS)等)に関与していることが報告されている。
*2 薬剤パーシスター:一定数の感受性菌を薬剤で処理しても、死滅せずに一部の菌が長期間生存する場合がある。このような、薬剤に低感受性を示す一群を薬剤パーシスターと呼ぶ。慢性感染症や感染症の再燃などに関連する。
*3 潜在性結核:結核菌に感染しているが、菌の休眠などにより結核を発病しない状態。
*4 高速原子間力顕微鏡(高速AFM):探針でなぞることでステージ上の物質の表面構造を画像化する解析装置。水溶液中の生体高分子(タンパク質、核酸など)の構造・動きをナノメーター、1秒以下の時空間解像度でタイムラプス撮影することが可能。
*5 粗子化分子動力学計算:分子動力学計算法の一種。高分子(タンパク質、核酸など)の構成単位(アミノ酸、塩基、リボースなど)を1粒子で表現した計算法。原子レベルの分子動力学計算に比べ計算過程が少なくて済むため、計算時間を短縮できる。複数の生体分子から構成される複合体の構造シミュレーションに適している。
本件に関するお問い合わせ先
【研究に関すること】
新潟大学大学院医歯学総合研究科細菌学分野
講師 西山 晃史(にしやま あきひと)
E-mail:anishi@med.niigata-u.ac.jp
京都大学複合原子力科学研究所粒子線基礎物性研究部門
助教 清水 将裕(しみず まさひろ)
E-mail:shimizu.masahiro.3n@kyoto-u.ac.jp
金沢大学ナノ生命科学研究所
教授 古寺 哲幸(こでら のりゆき)
E-mail:nkodera@staff.kanazawa-u.ac.jp
九州大学大学院薬学研究院創薬構造解析学分野
講師 眞胗 浩太(まやなぎ こうた)
E-mail:maya@phar.kyushu-u.ac.jp
【広報担当】
新潟大学広報事務室
E-mail:pr-office@adm.niigata-u.ac.jp
京都大学渉外部広報課国際広報室
E-mail:comms@mail2.adm.kyoto-u.ac.jp
金沢大学ナノ生命科学研究所事務室
E-mail:nanolsi-office@adm.kanazawa-u.ac.jp
九州大学広報課
E-mail:koho@jimu.kyushu-u.ac.jp